Présentation globale du complexe majeur d'histocompatibilité
- Présentation générale
- Structure des locus et des gènes MHC-I et MHC-II
- Structure des protéines MHC-I et MHC-II
- Structure générale des protéines MHC-I et MHC-II
- Structure des domaines G des protéines MHC-I et MHC-II
- Description
- Description du sillon
- Description des sites de liaison entre le peptide et le CMH
- Orientation du peptide dans le site d'ancrage
- Intéractions entre le MHC-I, la B2M, les peptides et le TR
- Prédiction des peptides susceptibles de se lier aux protéines CMH
- Intéractions entre les chaînes I-ALPHA (MHC-I) et la B2M ou I-ALPHA-like (MHC-I-like) et la B2M
- Complexe trimoléculaire TR/peptide/MHC
- Description
- Structure des domaines C-LIKE des protéines MHC-I et MHC-II
- Comparaison des séquences protéiques des différents gènes CMH
- Comparaison des séquences protéiques du G-DOMAIN
- IMGT unique numbering
- Protein display
- IMGT/Collier de Perles
- Comparaison des séquences protéiques du C-LIKE-DOMAIN
- IMGT unique numbering
- Protein display
- IMGT/Collier de Perles
- Comparaison des séquences protéiques du G-DOMAIN
- Complexité du CMH
- Fonctionnement du CMH
- MHC-I
- Etapes de l'expression à la surface cellulaire d'un complexe pMHC-Ia
- Voie de dégradation des protéines endogènes
- Voies de biosynthèse et de chargement en peptides des MHC-I
- Présentation des antigènes à la surface des cellules
- Cas particulier des cellules cancéreuses ou infectées par un pathogène
- Fonctionnement des protéines MHC-Ia et MHC-Ib
- La bêta-2-microglobuline (B2M)
- Etapes de l'expression à la surface cellulaire d'un complexe pMHC-Ia
- MHC-II
- MHC-I
- Bibliographie
I. Présentation générale
Introduction
Les molécules du complexe majeur d'histocompatibilité CMH (MHC en anglais pour major histocompatibility complex) sont nommées human leucocyte antigen (HLA) chez l'homme. Ces protéines transmembranaires appartiennent,
- par leur domaine de type G (pour groove ou sillon en français), à la superfamille des MhcSF
- et par leur domaine de type C (pour constant), à la superfamille des immunoglobulines (IgSF).
Les protéines CMH jouent un rôle clé dans la réponse immunitaire adaptative de tous les Gnasthostomata (vertébrés à mâchoires), en présentant des peptides issus de protéines endogènes ou exogènes aux récepteurs T (TR) des lymphocytes T (figure 1).
L'activation du lymphocyte T nécessite:
- tout d'abord une interaction du CD8 avec le MHC-I ou du CD4 avec le MHC-II,
- suivie des interactions entre le TR et le complexe peptide/MHC,
- et enfin de l'activation des co-récepteurs CD3.
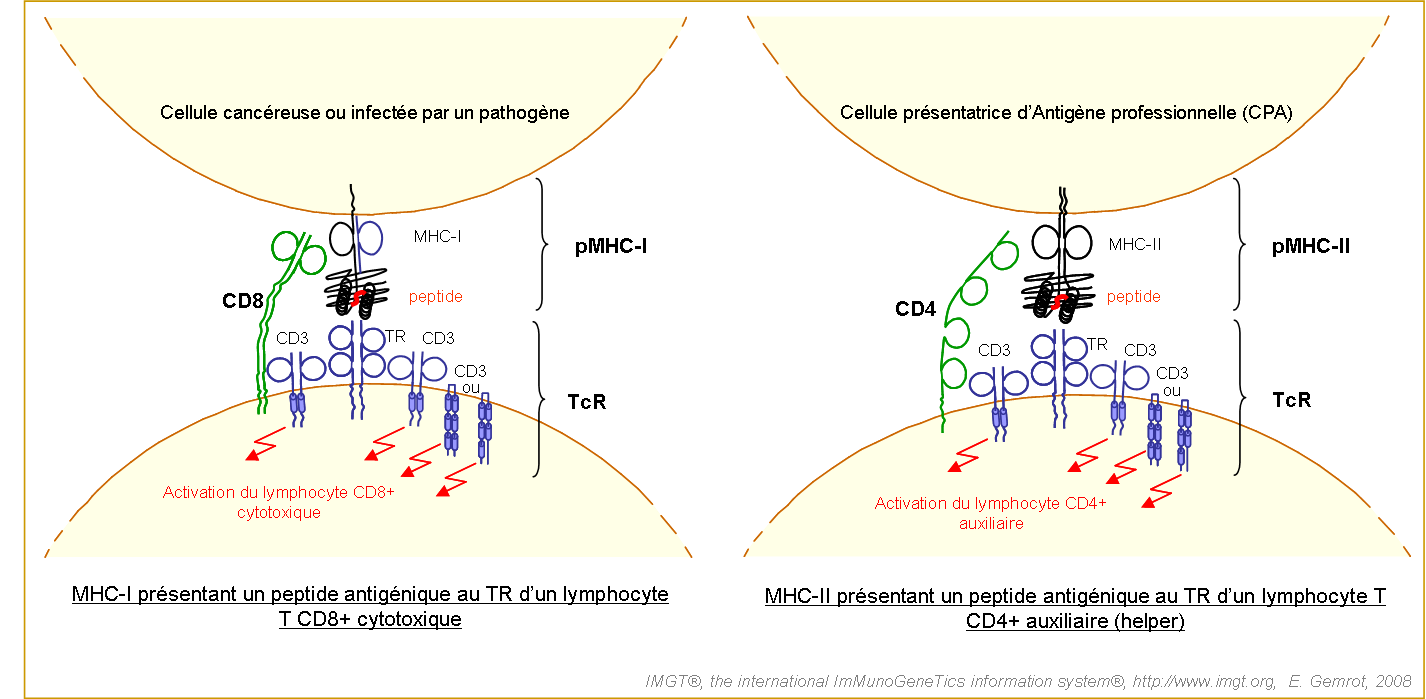 Figure 1. Représentation 2D des complexes trimoléculaires TR/peptide/MHC, des corécepteurs CD3 et des protéines associées CD4 et CD8 du lymphocyte T.
Figure 1. Représentation 2D des complexes trimoléculaires TR/peptide/MHC, des corécepteurs CD3 et des protéines associées CD4 et CD8 du lymphocyte T.
Le cas du MHC-I présentant un peptide antigénique au TR d'un lymphocyte T CD8+ cytotoxique est schématisé par la figure à gauche. Le cas du MHC-II présentant un peptide antigénique au TR d'un lymphocyte T CD4+ auxiliaire (ou helper) est schématisé par la figure à droite. Le récepteur T (TR) interagit avec le peptide mais aussi avec les domaines G-ALPHA-I et G-ALPHA-II du MHC-I ou avec les domaines G-ALPHA et G-BETA du MHC-II. pMHC correspond à l'association d'une proteine du complexe majeur d'histocompatibilité avec un peptide. TcR est situé à la surface des lymphocytes T. Il correspond à l'association du récepteur T avec les corécepteurs CD3. CD3 sont des corécepteurs participant à la transduction du signal de reconnaissance du pMHC au lymphocyte T. CD4 et CD8 sont des corécepteurs stabilisant les complexes TR/peptide/MHC en réalisant des liaisons avec la région constante C-LIKE-DOMAIN des CMH.
Les protéines CMH appartiennent à deux classes MHC-I et MHC-II, elles mêmes divisées en deux sous-classes (Lefranc et al. 2005a).
Tableau 1. Organisation des classes et des sous-classes des protéines du CMH :
| MHC-I | MHC-II |
|---|---|
| - Protéines CMH conventionnelles: MHC-Ia | - Protéines CMH conventionnelles: MHC-IIa |
| - Protéines CMH non conventionnelles: MHC-Ib | - Protéines CMH non conventionnelles: MHC-IIb |
ATTENTION : Les protéines du CMH conventionnelles sont souvent désignées MHC-I et MHC-II quand il n'y a pas de confusion possible avec les protéines du CMH non conventionnelles.
Il existe d'autres protéines qualifiées CMH de classe III (MHC-III). Elles ont été dénommées ainsi, en raison de la localisation de leurs gènes entre ceux des CMH de classe I et II, sur le bras court du chromosome 6 (6p21.3) chez l'homme (17p chez la souris). Les protéines MHC-III n'ont cependant ni la structure ni les fonctions des protéines MHC-I et MHC-II.
Parallèlement, il existe des protéines MHC-I-like dont la structure est proche des protéines du MHC-I mais dont les fonctions biologiques, les ligands (protéiques, peptidiques ou lipidiques) et la localisation des gènes divergent des CMH.
A. Description des protéines CMH conventionnelles (MHC-Ia et MHC-IIa)
Tableau 2. Comparaison des caractéristiques des protéines MHC-Ia et MHC-IIa :
| MHC-Ia | MHC-IIa | |
|---|---|---|
| Structure des protéines (figure 5) | 1 chaîne lourde transmembranaire (I-ALPHA, 44kDa) stabilisée par la protéine bêta-2-microglobuline (B2M, 11,5 kDa). Les deux chaînes sont associées de manière non covalente. | 2 chaînes transmembranaires (II-ALPHA, 31-34 kDa et II-BETA, 26-29 kDa) liées par des liaisons non covalentes. |
| Organisation structurale des chaînes | - La chaîne I-ALPHA est formée par 3 domaines extracellulaires : • 2 domaines polymorphiques de type G (groove): G-ALPHA1 [D1] et G-ALPHA2 [D2]: l'association des deux domaines G forme un sillon où peut se loger un peptide de 8 à 11 AA, généralement endogène (Lefranc et al. 2005a) ; • 1 domaine constant de type C: C-LIKE [D3] (Lefranc et al. 2005b). - La chaîne B2M est formée d'un seul domaine extracellulaire C-LIKE. |
Les chaînes II-ALPHA et II-BETA ont une structure semblable. Chaque chaîne est formée de 2 domaines extracellulaires : • 1 domaine polymorphique de type G: G-ALPHA [D1] pour la chaîne II-ALPHA ou G-BETA [D1] pour la chaîne II-BETA (Lefranc et al. 2005a) ; • 1 domaine constant de type C: C-LIKE [D2] (Lefranc et al. 2005b). L'association des domaines G des deux chaînes forme un sillon où peut se loger un peptide de 12 à 25 AA. |
| Localisations cellulaire et tissulaire | Sur les membranes de toutes les cellules nucléées. Il faut noter cependant des exceptions comme l'expression faible voir nulle du MHC-Ia au niveau des cellules du cerveau, des testicules et de la barrière foeto-maternelle (sauf en cas d'induction par les cytokines inflammatoires interféron gamma). |
Sur les membranes des cellules présentatrices d'antigènes (CPA) professionnelles: lymphocytes B, macrophages, monocytes neutrophiles et cellules dendritiques. |
| Fonction | Présentation de peptides endogènes aux lymphocytes T CD8+ cytotoxiques (figure 16). La protéine MHC-Ia interagit également avec différents récepteurs inhibiteurs des cellules NK appartenant aux familles multigéniques KIR (Snyder et al. 1999), LIR (Cosman et al. 1997) et NKG2 (Tormo et al. 1999) (figure 20). | Présentation de peptides exogènes issus de la protéolyse à pH acide de protéines endocytées aux lymphocytes T CD4+ auxiliaires (ou helper) (figure 21). |
| Peptides présentés | Peptides endogènes du soi et du non soi (peptides d'origine tumorale ou issus de pathogènes cytosoliques tels que les virus et les bactéries intracellulaires) majoritairement issus de la protéolyse par le protéasome, de protéines endogènes dénaturées ou inutilisées par la cellule (Arrigo et al. 1988). | Peptides d'origine extracellulaire (provenant de la lyse de protéines pathogènes, de cellules infectées, de cellules altérées ou du non soi endocytées par les CPA). |
| Gènes (Lefranc et al. 2005a) et localisation chromosomique | - 3 gènes chez l'homme: HLA-A, HLA-B et HLA-C (6p21.3). - 3 gènes principaux chez la souris: H2-K, H2-L et H2-D (17p18.43 à 17p19.3). |
- 7 à 11 gènes chez l'homme: HLA-DPA, HLA-DPB, HLA-DQA, HLA-DQB, HLA-DRA, et plusieurs HLA-DRB (2 à 5 suivant les haplotypes) (6p21.3). - 4 gènes fonctionnels chez la souris: H2-AA, H2-AB, H2-EA, H2-EB (17p18.64 à 17p18.70) et H2-P non productif (17p18.51). |
B. Description des protéines CMH non conventionnelles (MHC-Ib et MHC-IIb)
Les protéines CMH non conventionnelles ont de grandes similarités structurales avec les protéines CMH conventionnelles. Cependant, elles sont exprimées par des cellules distinctes et possèdent des fonctions différentes (Braud et al. 1999). En effet, elles ne présentent pas de peptides aux TR des lymphocytes T et ont des rôles qui leur sont propres.
Tableau 3. Comparaison des caractéristiques des protéines MHC-Ib et MHC-IIb :
| MHC-Ia | MHC-IIa | |
|---|---|---|
| Structure des protéines (figure 5) | 1 chaîne lourde transmembranaire (I-ALPHA, 44kDa) stabilisée par la protéine bêta-2-microglobuline (B2M, 11,5 kDa). Les deux chaînes sont associées de manière non covalente. | 2 chaînes transmembranaires (II-ALPHA, 31-34 kDa et II-BETA, 26-29 kDa) liées par des liaisons non covalentes. |
| Organisation structurale des chaînes | - La chaîne I-ALPHA est formée par 3 domaines extracellulaires : • 2 domaines polymorphiques de type G (groove): G-ALPHA1 [D1] et G-ALPHA2 [D2](Lefranc et al. 2005a) ; • 1 domaine constant de type C: C-LIKE [D3] (Lefranc et al. 2005b). - La chaîne B2M est formée d'un seul domaine extracellulaire C-LIKE. |
Les chaînes II-ALPHA et II-BETA ont une structure semblable. Chaque chaîne est formée de 2 domaines extracellulaires : • 1 domaine polymorphique de type G: G-ALPHA [D1] pour la chaîne II-ALPHA ou G-BETA [D1] pour la chaîne II-BETA (Lefranc et al. 2005a) ; • 1 domaine constant de type C: C-LIKE [D2] (Lefranc et al. 2005b). |
| Localisations cellulaire et tissulaire | - La protéine HLA-G est localisée dans la sueur, les larmes, les urines, la bile, les sérums et le lait ; mais aussi, à l'interface foeto-maternelle (Xiaohua et al. 1990). - Les protéines HLA-E et HLA-G sont localisées dans les cellules trophoblastiques. - La protéine HLA-F est localisée dans les cellules T, les amygdales, la rate, le thymus (Lepin et al. 2000)... |
- Les protéines du MHC-IIb sont absentes de la surface cellulaire. Elles sont situées au niveau des compartiements endosomes/lysosomes (Karlsson 2005). - Les protéines HLA-DMA et HLA-DMB sont essentiellement présentes dans les vésicules intracytoplasmiques (Karlsson 2005). - Les protéines HLA-DOA et HLA-DOB sont principalement retrouvées dans les cellules B et les cellules épithéliales du thymus (Karlsson 2005). |
| Fonctions spécifiques | - Les protéines MHC-Ib protègent les cellules saines de la lyse par les cellules cytotoxiques NK et T CD8+: • la protéine HLA-E inhibe ou active l'activité cytolytique des cellules cytotoxiques natural killer (NK) suivant les récepteurs de la cellule NK avec lesquels il interagit: CD94/NKG2C (activation) CD94/NKG2A (inhibition) ; • la protéine HLA-F interagit entre autres avec les récepteurs LIR1 (ILT2) et LIR2 (ILT4) afin de modifier le seuil d'activation des cellules immunitaires effectrices (Lepin et al. 2000) ; • la protéine HLA-G inhibe l'activité cytolytique des cellules cytotoxiques et a un rôle protecteur contre les rejets et les infections du placenta. - Certaines protéines MHC-Ib solubles induisent l'apoptose des cellules cytotoxiques T CD8+. |
- Les protéines HLA-DMA et HLA-DMB ne fixent pas de peptides dans leur sillon. Par contre, ces protéines jouent un rôle dans le démentellement du complexe CLIP/MHC-II, permettant ainsi le chargement des peptides antigèniques dans le sillon des MHC-IIa. De plus, elles stabilisent les complexes pMHC-II et contribuent à leur adressage vers la surface des cellules hôtes (figure 21). - HLA-DOA et HLA-DOB sont des protéines dont la fonction est pH dépendant. De part cette propriété, elles peuvent inhiber le chargement des peptides dans les endosomes précoces ou le favoriser, pour certains peptides, dans les endosomes matures (Karlsson 2005). |
| Particularités | - Les protéines MHC-Ib membranaires peuvent être clivées par des métalloprotéinases. Il en résulte des protéines de MHC-Ib solubles (ex HLA-G). - Augmentation de la concentration en MHC-I solubles dans certains cas de tumeurs. |
- Les protéines MHC-IIb membranaires peuvent être clivées par des métalloprotéinases. Il en résulte des protéines MHC-IIb solubles (ex MHC-DO et MHC-DM). - Les protéines HLA-DOA et HLA-DOB nouvellement synthétisées sont retrouvées uniquement en association avec HLA-DM. Cette association permet de stabiliser les protéines HLA-DO et permet leur sortie du réticulum endoplasmique (Liljedahl et al. 1996). Par ailleurs, il n'a pas encore été établit si le complexe MHC-DO/DM interagit avec les protéines du MHC-IIa ou si les protéines HLA-DM et HLA-DO se dissocient avant l'intéraction. Plus récemment, il a été montré que la protéine HLA-DO peut s'associer à la protéine du MHC-I libre pour former des complexes qui peuvent être détectées en petites quantités à la surface cellulaire (Van Lith et al. 2003), mais la fonction de ces complexes n'est pas encore connue. |
| Gènes (Lefranc et al. 2005a) et localisation chromosomique | - 3 gènes fonctionnels chez l'homme: HLA-E, HLA-F et HLA-G (6p21.3). - 2 pseudogènes chez l'homme: HLA-H et HLA-J (ou X). - 3 groupes de gènes chez la souris: H2-Q, H2-M et H2-T (17p19.14 à 17p20.43). |
- 4 gènes chez l'homme: HLA-DMA, HLA-DMB, HLA-DOA et HLA-DOB (6p21.3). - 4 gènes chez la souris: H2-DMA, H2-DMB, H2-DOA et H2-DOB (17p18.52 à 17p18.58). |
C. Description des protéines du MhcSF autres que CMH (MHC-I-like)
Tableau 4. Caractéristiques des protéines MHC-I-like :
| Structure des G-REGION | Différente de celle des MHC-Ia et MHC-Ib, même si elle reste assez proche comme pour le cas du gène CD1 (chaîne alpha glycosylée + protéine B2M) |
|---|---|
| Fonctions spécifiques | - AZGP1 (Zinc-alpha-2-glycoprotein 1): régule la dégradation des acides gras dans les adipocytes en se fixant sur des dérivés lipidiques (Sanchez et al. 1999). - Famille CD1: présente des antigènes lipidiques de types phospholipides (glycérolipides et Dolichol lipides) ou glycosphingolipides aux TR alpha-bêta et gamma-delta des lymphocytes T et des cellules natural killer (NK) dans le but de réguler la réponse immunitaire et la réponse anti-tumorale (Zeng et al. 1997). - EPCR (endothelial cell protein C receptor): interagit avec des dérivés lipidiques ainsi que les protéines C et est impliqué dans la voie de coagulation du sang (Simmonds et al. 1999). - FCRN (neonatal Fc receptor) ou FCRT: transporte les Ig à travers le placenta et régit l'immunité néonatale (West et al. 2000). - HFE (hemochromatosis): prend part à l'homéostasie du fer en interagissant avec le récepteur transferrine TfR1 (Feder et al. 1998). - Famille MIC: est induit par le stress lié aux tumeurs et infections virales ; il assure la reconnaissance et la destruction des cellules infectées et tumorales par le système immunitaire en interagissant avec le récepteur NKG2D des cellules NK (Holmes et al. 2002, Frigoul et al. 2005). - MR1 (MHC-related 1) : fonction inconnue (Miley et al. 2003). - Famille RAE (retinoid acid early): stimule la production de cytokine/chemokine et l'activité cytotoxique des cellules NK en interagissant avec le récepteur NKG2D (Li et al. 2002). |
| Gènes (Lefranc et al. 2005a) et localisation chromosomique | - Homo sapiens: AZGP1 (7q22), famille CD1 (A, B, C, D, E) (1q22-q23), EPCR (20q11.2), FCGRT (19q13.3), HFE (6p22.1), famille MIC (A et B) (6p21.3), MR1 (1q25.3), famille RAE (T1E, T1H, T1I, T1L, T1N) (6q24.2 à 6q25.3). - Mus musculus: AZGP1, CD1D1, EPCR, FCGRT, HFE (chr. 13), MR1 (chr. 1) RAE (1B et 1G) (chr.10). |
II. Structure des locus et des gènes MHC-I et MHC-II
A. Organisation génomique des locus CMH
Les régions génomiques des locus CMH sont denses en gènes. On retrouve certains des gènes dupliqués associés ou non à d'autres gènes de régulation (TAP, PSMB8, PSMB9, ABCB3B...) (Kulski et al. 2002).
Tableau 5. Correspondance des différentes nomenclatures des gènes de régulation humains du CMH :
| Nomenclature IMGT | Alias | Localisation chromosomique | Noms des gènes approuvés par HGNC |
|---|---|---|---|
| TAP1 | ABCB2, PSF1, RING4, D6S114E | 6p21.3 | Transporter 1, ATP-binding cassette, sub-family B (MDR/TAP) |
| ABCB3 | TAP2, PSF2, RING11, D6S217E | 6p21.3 | Transporter 2, ATP-binding cassette, sub-family B (MDR/TAP) |
| PSMB8 | RING10, D6S216E, PSMB5i, beta5i | 6p21.3 | Proteasome (prosome, macropain) subunit, beta type, 8 (large multifunctional peptidase 7) |
| PSMB9 | RING12, beta1i, PSMB6i | 6p21.3 | Proteasome (prosome, macropain) subunit, beta type, 9 (large multifunctional peptidase 2) |
| PSMB10 | LMP10, MGC1665, beta2i | 6q22.1 | Proteasome (prosome, macropain) subunit, beta type, 10 |
L'organisation multigénique des locus CMH est différente selon les espèces (Clark et al. 2001, Matsuo et al. 2002, Michalova et al. 2000, Sambrook et al. 2005, Shiina et al. 2004, Shiina et al. 2005 et Tsukamoto et al. 2005). Ainsi, les gènes du CMH peuvent :
- se situer sur un ou plusieurs chromosomes et dans des locus différents (tableau 6) ;
- présenter ou non une co-ségrégation entre les gènes MHC-I et MHC-II ;
- être associés ou non à d'autres gènes ;
- avoir des tailles d'introns variables (ex : l'intron 2 du gène UBA de la truite arc-en-ciel (MHC-I) atteint 18kb alors qu'il n'est composé que de quelques centaines de paires de bases pour les MHC-I des autres vertébrés) (Shiina et al. 2005).
L'organisation schématique des locus CMH est représentée dans la figure 2 « Comparison of the human, Atlantic salmon, rainbow trout, medaka, zebrafish and fungu MHC class I » de la référence Lukacs et al. 2007.
Tableau 6. Localisation des locus CMH :
| Espèce | Type de CMH | Localisation chromosomique |
Position (en cM) | Positions NCBI map viewer (pb) | Accession number |
|---|---|---|---|---|---|
| Homme (Homo sapiens) |
MHC-I | 6p | 21.3 | 29799096-33389967 | (map viewer) |
| MHC-II | 2693616-3352449 | ||||
| MHC-III | |||||
| Souris (Mus musculus) |
MHC-I | 17p | 18.40 - 20.43 | 341172680-37620474 | (map viewer) |
| MHC-II | 18.51 - 18.70 | 34221558-34481588 | |||
| Truite arc-en-ciel (Oncorhynchus mykiss) |
MHC-I | 18q (a) 14q |
18q21 14q12 |
AB162342 AB162343 |
|
| MHC-II | 17p (b) | ||||
| Saumon (Salmo salar) |
MHC-I | 27p (c) | EF427381, EF441211, EF427384, EF210363 et EF427383 | ||
| MHC-II | 14 | EF427379, EF427382, EF427378 et EF427377 |
B. Organisation structurale des exons des gènes MHC-I et MHC-II et correspondance avec les domaines protéiques
De manière générale, les gènes CMH appartiennent à des familles multigéniques et multialléliques (pour le MHC-I) qui ont des tailles d'exons quasi similaires quelle que soit l'espèce.
L'organisation structurale des gènes CMH et leur correspondance avec les domaines protéiques peuvent être schématisés de la manière suivante:
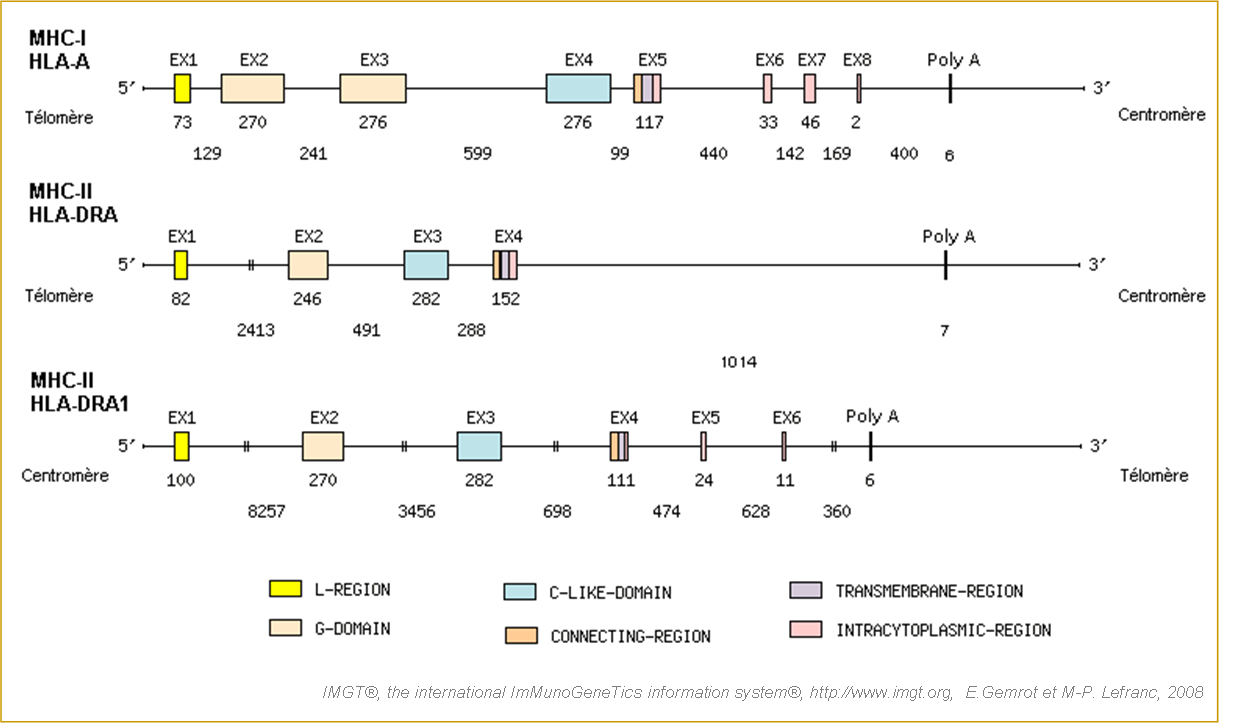 Figure 2. Structure des gènes humains HLA-A (MHC-Ia), HLA-DRA et HLA-DBR1 (MHC-IIa) présents sur le chromosome 6p21.3. (Lefranc et al. 2005a).
Figure 2. Structure des gènes humains HLA-A (MHC-Ia), HLA-DRA et HLA-DBR1 (MHC-IIa) présents sur le chromosome 6p21.3. (Lefranc et al. 2005a).
Gène HLA-A codant la chaîne I-ALPHA de la protéine HLA-A (MHC-Ia) de l'humain. Gène HLA-DRA codant la chaîne II-ALPHA de la protéine HLA-DRA (MHC-IIa) de l'humain. Gène HLA-DRB codant la chaîne II-BETA de la protéine HLA-DRB1 (MHC-IIa) de l'humain. Les gènes HLA-A et HLA-DRA sont forward tandis que le gène HLA-DRB1 est en revers sur le chromosome. La taille des introns et des exons est exprimée en paires de bases. Le signe (-||-) signifie que l'intron n'est pas à l'échelle. EMBL/GenBank/DDBJ accession numbers : HLA-A*0201 (K02883), HLA-DRA*0101 (J00203 et J00204) et HLA-DRB1*1402 (AL137064).
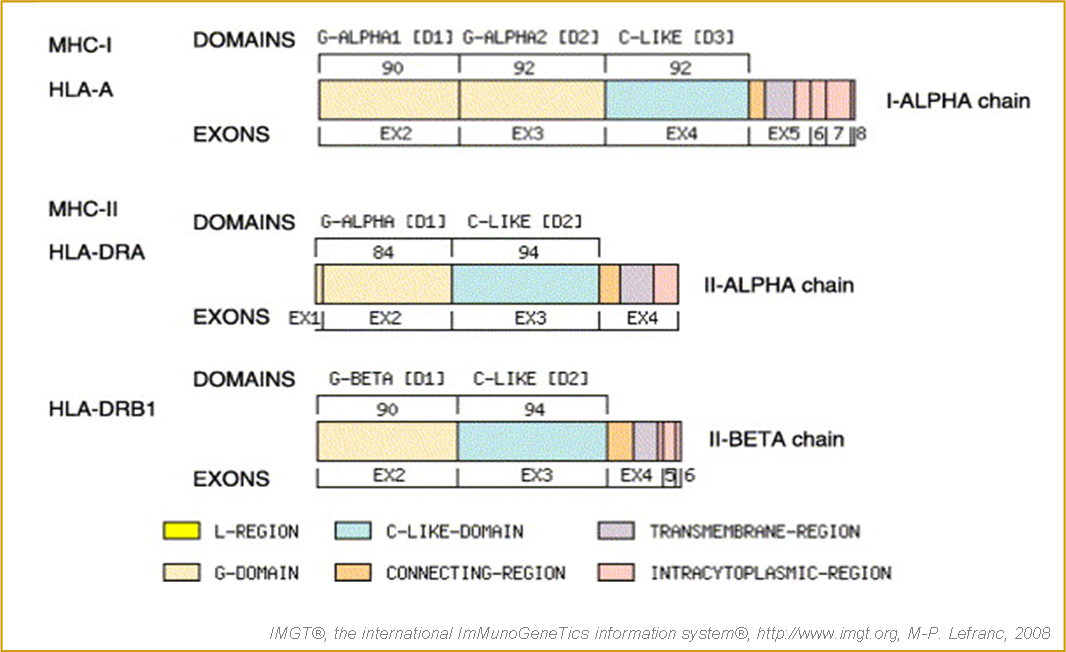 Figure 3. Correspondance entre exons et domaines pour les gènes humains HLA-A (MHC-Ia), HLA-DRA et HLA-DRB (MHC-IIa) (Lefranc et al. 2005a).
Figure 3. Correspondance entre exons et domaines pour les gènes humains HLA-A (MHC-Ia), HLA-DRA et HLA-DRB (MHC-IIa) (Lefranc et al. 2005a).
La taille des domaines est exprimée en nombre d'acides aminés. EMBL/GenBank/DDBJ accession numbers : HLA-A*0201 (K02883), HLA-DRA*0101 (J00203 et J00204) et HLA-DRB1*1402 (AL137064).
III. Structure des protéines MHC-I et MHC-II
A. Structure générale des protéines MHC-I et MHC-II
Les structures tridimensionnelles des protéines MHC-I (Bjorkman et al. 1985) et MHC-II (Brown et al. 1993) sont assez semblables bien que la structure des chaînes qui les constitue soit différente.
Ainsi, les protéines MHC-I sont constituées d'une chaîne lourde transmembranaire I-ALPHA associée de façon non covalente à la chaîne bêta-2-microglobuline (B2M). La chaîne I-ALPHA comprend trois domaines extracellulaires : deux G-DOMAIN (G-ALPHA1 [D1], G-ALPHA2 [D2]) (Lefranc et al. 2005a) et un C-LIKE-DOMAIN [D3] (Lefranc et al. 2005b) (figures 4 et 5).
Les protéines MHC-II sont constituées de deux chaînes, II-ALPHA et II-BETA, chacune comprenant un G-DOMAIN (G-ALPHA [D1] ou G-BETA [D1]) (Lefranc et al. 2005a) et un C-LIKE-DOMAIN [D2] (Lefranc et al. 2005b) (figures 4 et 5).
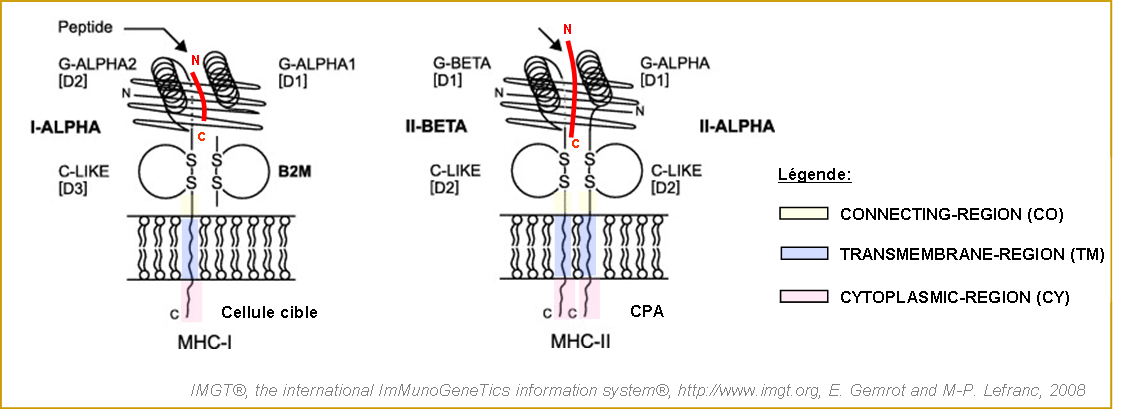 Figure 4. Représentation de la structure des protéines MHC-I et MHC-II (Kaas et al. 2004 et Lefranc et al. 2005a).
Figure 4. Représentation de la structure des protéines MHC-I et MHC-II (Kaas et al. 2004 et Lefranc et al. 2005a).
Le peptide antigénique est représenté par un trait rouge.
Les lettres N et C indiquent respectivement les extrémités N-terminale et C-terminale.
Dans le cas du MHC-I, l'extrémité N-terminale du peptide est ancrée dans le sillon.
Dans le cas du MHC-II, l'extrémité N-terminale du peptide est libre.
CPA signifie cellule présentatrice d'antigène professionnelle.
S-S correspond au pont disulfure.
[D1], [D2] et [D3] indiquent les domaines extracellulaires des chaînes protéiques numérotés à partir de l'extrémité N-terminale des chaînes.
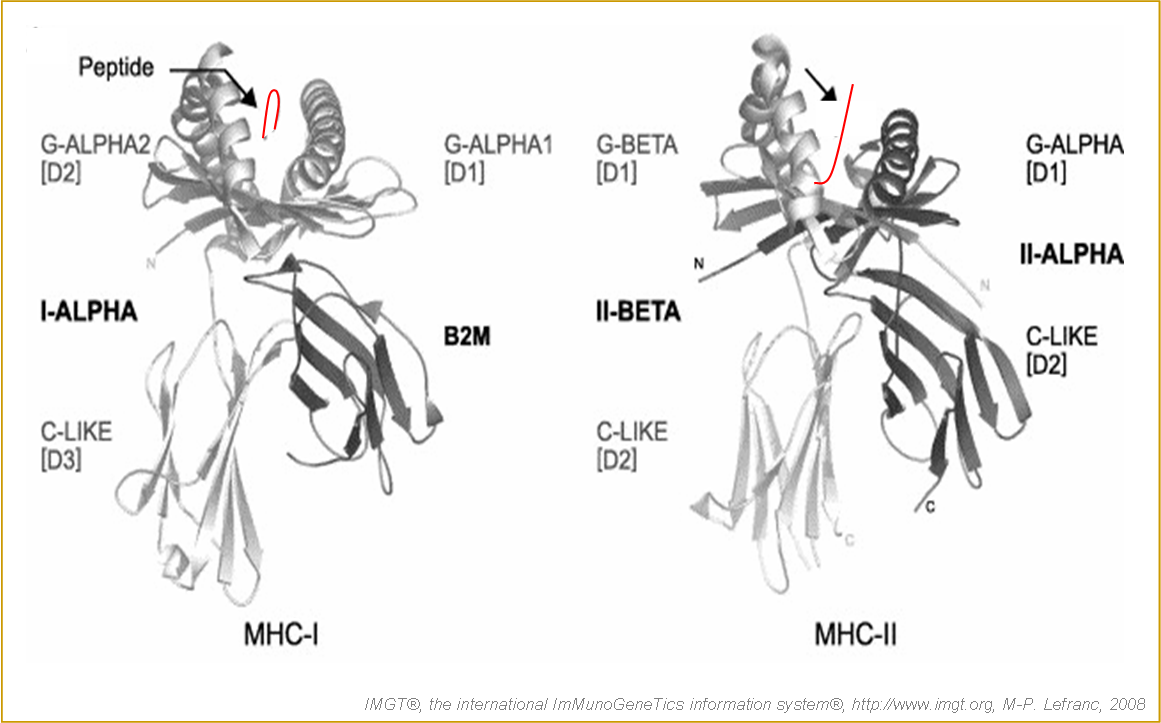 Figure 5. Représentation de la structure cristallographique des protéines MHC-I et MHC-II (Berman et al. 2000).
Figure 5. Représentation de la structure cristallographique des protéines MHC-I et MHC-II (Berman et al. 2000).
Les domaines et les chaînes sont décrits selon IMGT (Kaas et al. 2004 et Lefranc et al. 2005a). Le peptide antigénique est représenté par un trait rouge. [D1], [D2] et [D3] indiquent les domaines extracellulaires des chaînes protéiques numérotés à partir de l'extrémité N-terminale des chaînes. IMGT/3Dstructure-DB et PDB: MHC-I (1oga) et MHC-II (1j8 h).
B. Structure des domaines G des protéines MHC-I et MHC-II
1. Description
Chaque G-DOMAIN est constitué d'un feuillet de 4 brins bêta antiparallèles (A, B, C et D) et d'une longue hélice alpha.
Des analyses de polymorphisme des CMH montrent que les positions des acides aminés (AA) les plus variables se situent dans les G-DOMAIN et plus particulièrement au niveau des AA en contact avec les peptides antigèniques et les TR.
- Description du sillon
Le site de liaison du peptide antigénique a la forme d'un sillon. Il est constitué par l'association de deux G-DOMAIN. Le sillon est donc composé d'un plancher de 8 brins antiparallèles bêta sur lesquels reposent deux hélices alpha arrangées symétriquement (parois du sillon) (figure 6). Les hélices forment un angle de 40° avec les brins du feuillet bêta (Kaas et al. 2004).
Le sillon du MHC-I est constitué par les domaines G-ALPHA1 [D1] et G-ALPHA2 [D2] tandis que le sillon du MHC II est constitué par les domaines G-ALPHA [D1] de la chaîne II-ALPHA et G-BETA [D1] de la chaîne II-BETA (figure 6).
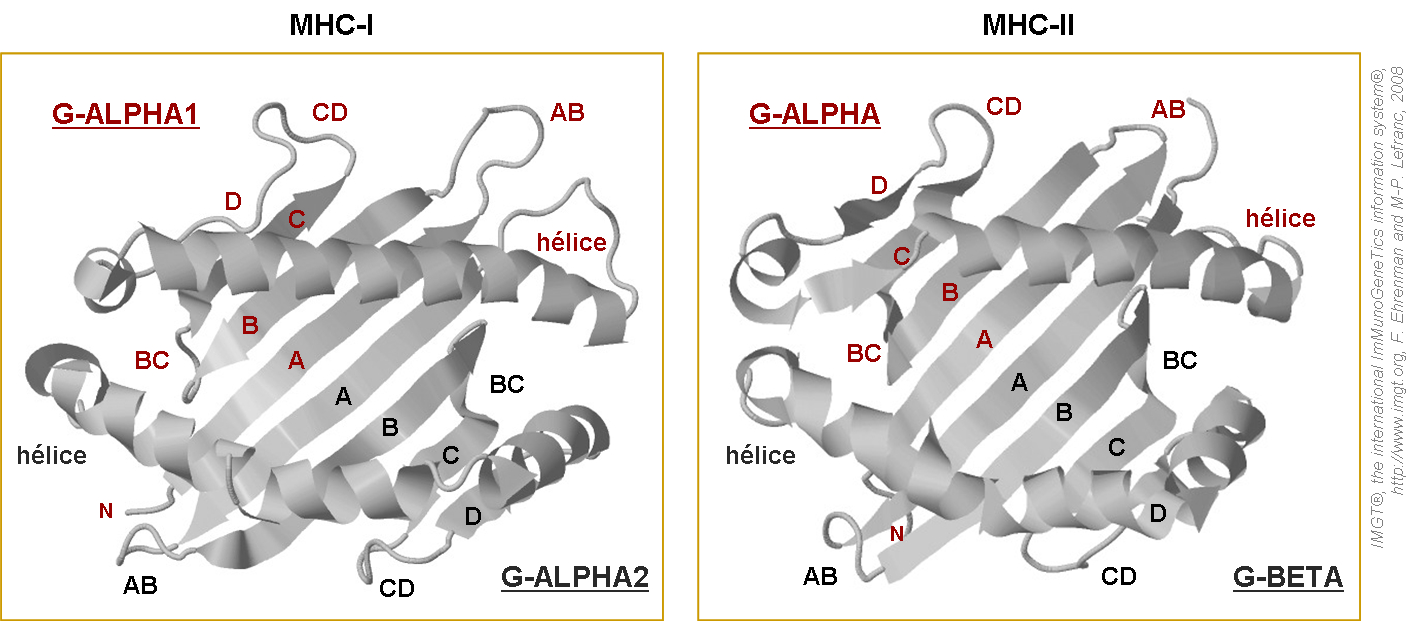 Figure 6. Sillons des protéines MHC-I et MHC-II.
Figure 6. Sillons des protéines MHC-I et MHC-II.
Les brins antiparallèles (A, B, C et D) sont liés entre eux par des boucles (respectivement AB, BC et CD). Les lettres rouges indiquent les brins et les boucles des domaines G-ALPHA1 (MHC-I) ou G-ALPHA (MHC-II). Les lettres noires indiquent les brins et les boucles des domaines G-ALPHA-2 (MHC-I) ou G-BETA (MHC-II). IMGT/3Dstructure-DB et PDB: MHC-I (1ao7_A) et MHC-II (1j8 h).
- Description des sites de liaison entre le peptide et le CMH
Le sillon du MHC-I est relativement fermé au niveau de ses extrémités. Il mesure 25A de long contre 10A de large et peut être lié à des peptides antigéniques de 8 à 11 AA. Les peptides antigéniques présentés ont leurs deux extrémités N et C terminales ancrées dans les repliements profonds A et F du sillon (ou pMHC contact site) tandis que leur partie centrale peut éventuellement sortir du site de liaison (Bjorkman et al. 1990) (figures 5 et 7).
Le sillon du MHC-II est quant à lui plus largement ouvert au niveau de ses extrémités. Il peut être lié à des peptides antigéniques de 12 à 25 AA. Cependant, seul 9 AA de ces peptides sont logés dans le sillon, les autres dépassant des extrémités (Brown et al. 1993). La position du peptide est plus ou moins horizontale dans le sillon (figures 5 et 7).
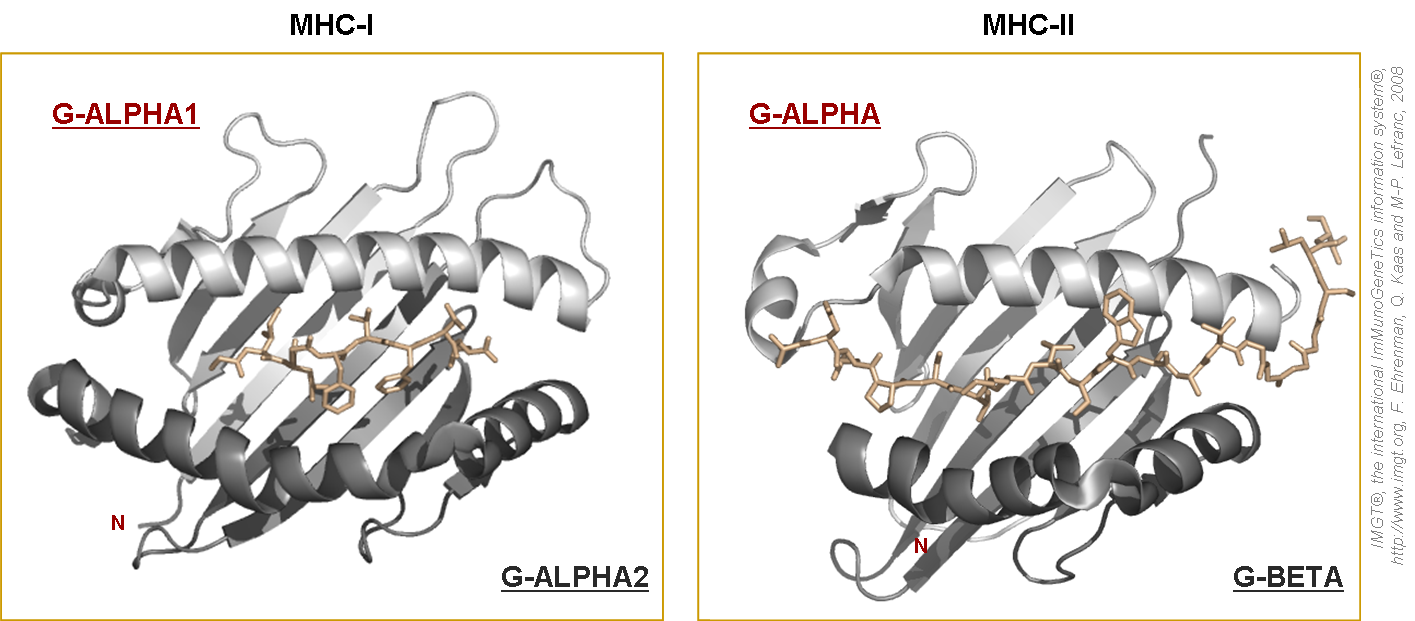 Figure 7. Sillons des protéines MHC-I et MHC-II présentant des peptides.
Figure 7. Sillons des protéines MHC-I et MHC-II présentant des peptides.
Les différences structurales entre les sillons des protéines MHC-I et MHC-II conditionnent ainsi la taille des peptides antigéniques présentés, mais aussi, le nombre et la localisation des liaisons entre les acides aminées du sillon et ceux des antigènes (Rammensee 1995). Ces sites de liaison peptidique sont également nommés « sites de contact » ou « poche d'ancrage » (figure 8 et tableau 7).
La stabilité du complexe pMHC fait intervenir ces sites de contact mais aussi d'autres liaisons intermédiaires.
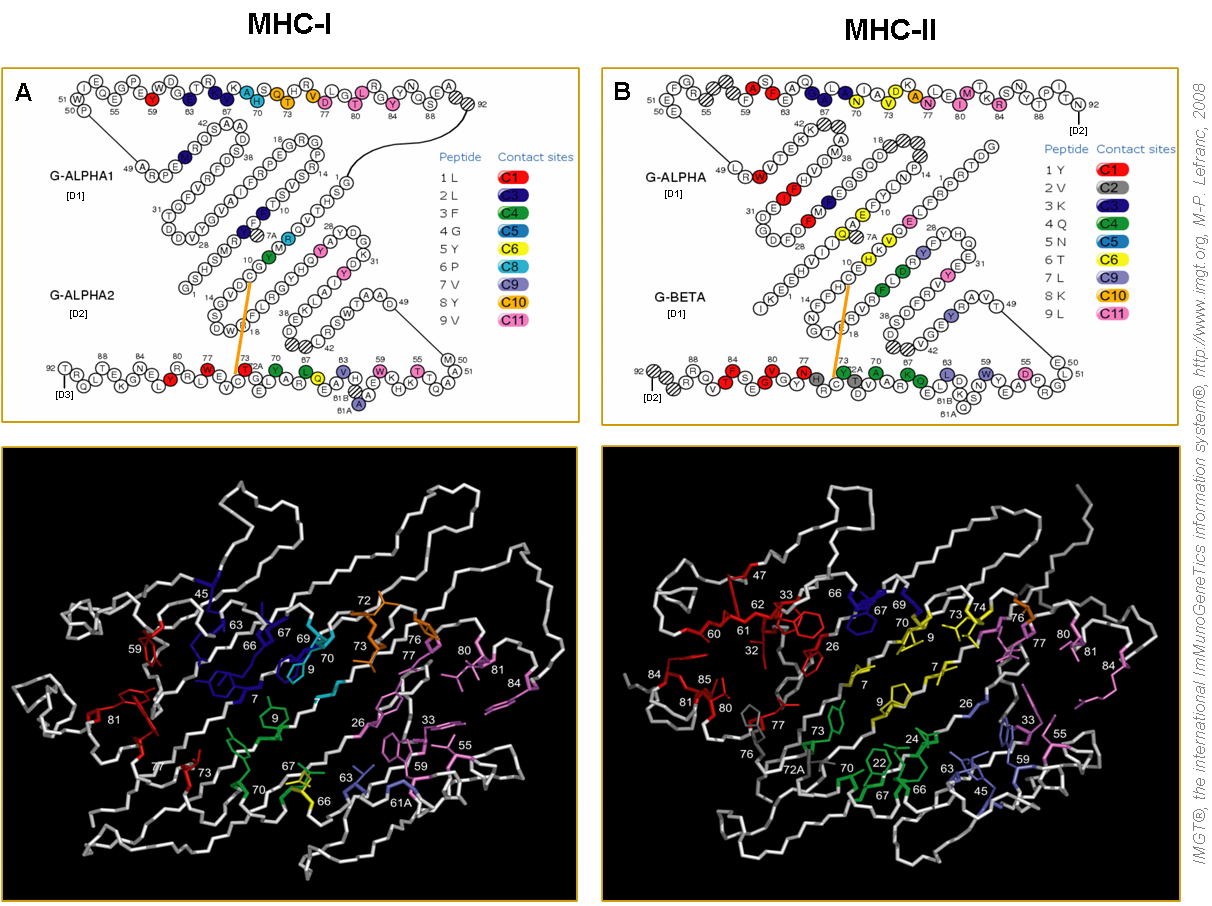 Figure 8. Représention des sites de contact IMGT entre le peptide et les domaines G du CMH (Kaas et al. 2004).
Figure 8. Représention des sites de contact IMGT entre le peptide et les domaines G du CMH (Kaas et al. 2004).
Les ponts disulfures du G-ALPHA2 (MHC-I) et du G-BETA (MHC-II) se trouvent entre les cystéines C11 et C74 et sont symbolisés par les traits orange. IMGT/3Dstructure-DB et PDB: MHC-I (1ao7) et MHC-II (1j8h).
La numérotation unique des domaines G (Lefranc et al. 2005a) a permis de standardiser l'analyse des contacts entre peptides et les CMH, mais aussi de définir 11 sites de contact IMGT (Kaas et al. 2004). Ces sites sont déterminés à partir des données structurales introduites dans IMGT/3Dstructure-DB et sont désignés « C1 à C11 » (figure 8).
Tableau 7. Sites de contact pMHC d'IMGT :
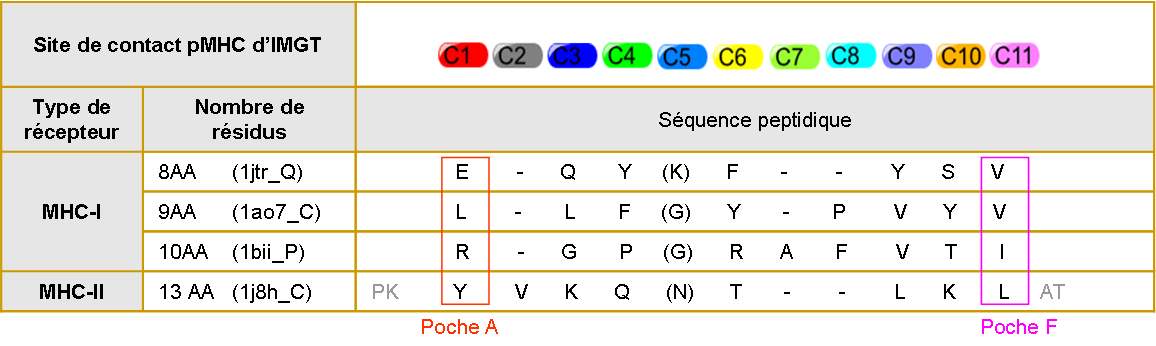 Les lettres entre parenthèses correspondent aux acides aminés uniquement en contact avec le récepteur T. Les lettres grises correspondent aux acides aminés qui dépassent du sillon. Les tirets correspondent à des gaps. Le cadre rouge correspond à la poche A et le cadre violet correspond à la poche F des sillons. Les positions des poches A et F du MHC-II ont été déterminées à partir de données statistiques car les sites de contact C1 et C11 des MHC-II sont relativement mal définis. Le site de contacte C2 caractérise les protéines MHC-II. Le site de contact C7 caractérise un peptide de 10AA lié à la protéine MHC-I. De la même manière, le site de contact C8 caractérise un petide de 9AA ou 10AA lié à la protéine MHC-I.
Les lettres entre parenthèses correspondent aux acides aminés uniquement en contact avec le récepteur T. Les lettres grises correspondent aux acides aminés qui dépassent du sillon. Les tirets correspondent à des gaps. Le cadre rouge correspond à la poche A et le cadre violet correspond à la poche F des sillons. Les positions des poches A et F du MHC-II ont été déterminées à partir de données statistiques car les sites de contact C1 et C11 des MHC-II sont relativement mal définis. Le site de contacte C2 caractérise les protéines MHC-II. Le site de contact C7 caractérise un peptide de 10AA lié à la protéine MHC-I. De la même manière, le site de contact C8 caractérise un petide de 9AA ou 10AA lié à la protéine MHC-I.
- Orientation du peptide dans le site d'ancrage
L'orientation a été sujette à discussions lors des premières publications des structures 3D de pMHC. En effet, des études théoriques ont montré que les peptides pourraient se lier dans les deux sens (Gopalakrishnan et al. 1992). Toutefois, seule une orientation a été observée expérimentalement : montrant une extrémité N-terminale peptidique proche de l'extrémité N-terminale de l'hélice G-ALPHA1 pour les MHC-I et de l'hélice G-ALPHA pour les MHC-II (au fond sur les figures 4 et 5 et à gauche sur les figures 6, 7 et 8).
2. Intéractions entre le MHC-I, la B2M, les peptides et le TR
- Prédiction des peptides susceptibles de se lier aux protéines CMH
Les sites de liaison des CMH de classe I et II ont une structure similaire et certains points communs dans la présentation des peptides (Brown et al. 1993). Ainsi, les sites d'ancrage sont tapissés d'acides aminés polymorphes qui vont déterminer, de par leurs propriétés physio-chimiques, aussi bien la sélectivité que la spécificité du CMH vis-à-vis des peptides antigéniques.
L'alignement des séquences des peptides et les structures 3D de pMHC révèle que l'ancrage de certaines positions du peptide détermine la liaison de ce peptide à des allèles particuliers de CMH. Basés sur ces observations, les outils de prédictions des peptides susceptibles de se lier aux protéines MHC-I ont été développés.
Ces outils peuvent être classés en trois grands groupes : les techniques basées sur les alignements de séquences de peptides, celles basées sur l'alignement d'une séquence de peptide sur une structure pMHC (threading) et enfin celles s'apuillant sur la constuction de la structure 3D du complexe peptide/MHC. Ces prédictions permettrent d'obtenir différents niveaux de détails : (1) la prédiction binaire (liaison ou non au CMH), (2) la prédiction de groupes discrets d'affinité, (3) et la prédiction de l'énergie libre d'intéraction.
En ce qui concerne le MHC-II, la majorité des sites de fixation des peptides se fait au niveau de motifs peptidiques peu spécifiques (Falk et al. 1991). De ce fait, contrairement aux protéines MHC-I, il est difficile de donner une prédiction des peptides susceptibles de se lier aux protéines MHC-II. Bien que des outils aient été développés, leur performance reste nettement inférieure à ceux des prédictions de CMH de classe I.
- Intéractions entre les chaînes I-ALPHA (MHC-I) et la B2M ou I-ALPHA-like (MHC-I-like) et la B2M
Les positions des acides aminés des G-DOMAIN des MHC-I et des G-LIKE-DOMAIN des MHC-I-like entrant dans l'interaction avec le C-LIKE-DOMAIN de la B2M diffèrent selon le type de récepteur (CD1, FCGRT et HFE). Cependant, huit positions des domaines G-ALPHA1 et G-ALPHA1-LIKE [D1] et 11 positions des domaines G-ALPHA2 et G-ALPHA2-LIKE [D2] semblent être impliquées dans l'association quelque soit le type de MhcSF (Duprat et al. 2006).
Ainsi, pour les domaines G-ALPHA1 et G-ALPHA1-LIKE, les positions des acides aminés responsables de l'interaction avec la B2M se situent au niveau des brins A, B, C et D et des boucles AB et BC.
Pour les domaines G-ALPHA2 et G-ALPHA2-LIKE, les positions des acides aminés responsables de l'interaction avec la B2M se situent au niveau des brins A, B et C et de la boucle BC.
Il est à remarquer que chaque position correspond à des acides aminés aux propriétés physico-chimiques similaires.
- Complexe trimoléculaire TR/peptide/MHC
Plusieurs études ont tenté de déterminer les règles globales de la formation des complexes TR/peptide/MHC (Vasmatzis et al. 1996 et Kjer-Nielsen et al. 2003) néanmoins, aucun principe permettant de prédire l'orientation précise du TR n'a été découvert (Stewart-Jones et al. 2003).
Le TR s'oriente de façon diagonale par rapport au peptide mais l'angle est variable et sa position par rapport aux extrémités l'est aussi (Teng et al. 1998). De manière générale, les positions du TR par rapport au CMH varient d'un cas sur l'autre.
Les CDR et la boucle AB du TR réalisent des liaisons avec les hélices des CMH (figure 9).
- Ainsi, le CDR2 du V-BETA se place au niveau de l'hélice du MHC-I G-ALPHA1 ou du MHC-II G-ALPHA
- alors que le CDR2 du V-ALPHA se place au niveau de l'hélice du MHC-I ALPHA2 ou du MHC-II G-BETA.
Les interactions entre les chaînes du récepteur T et du CMH suggèrent que le TR est aussi spécifique du CMH que du peptide (Kaas et al. 2004).
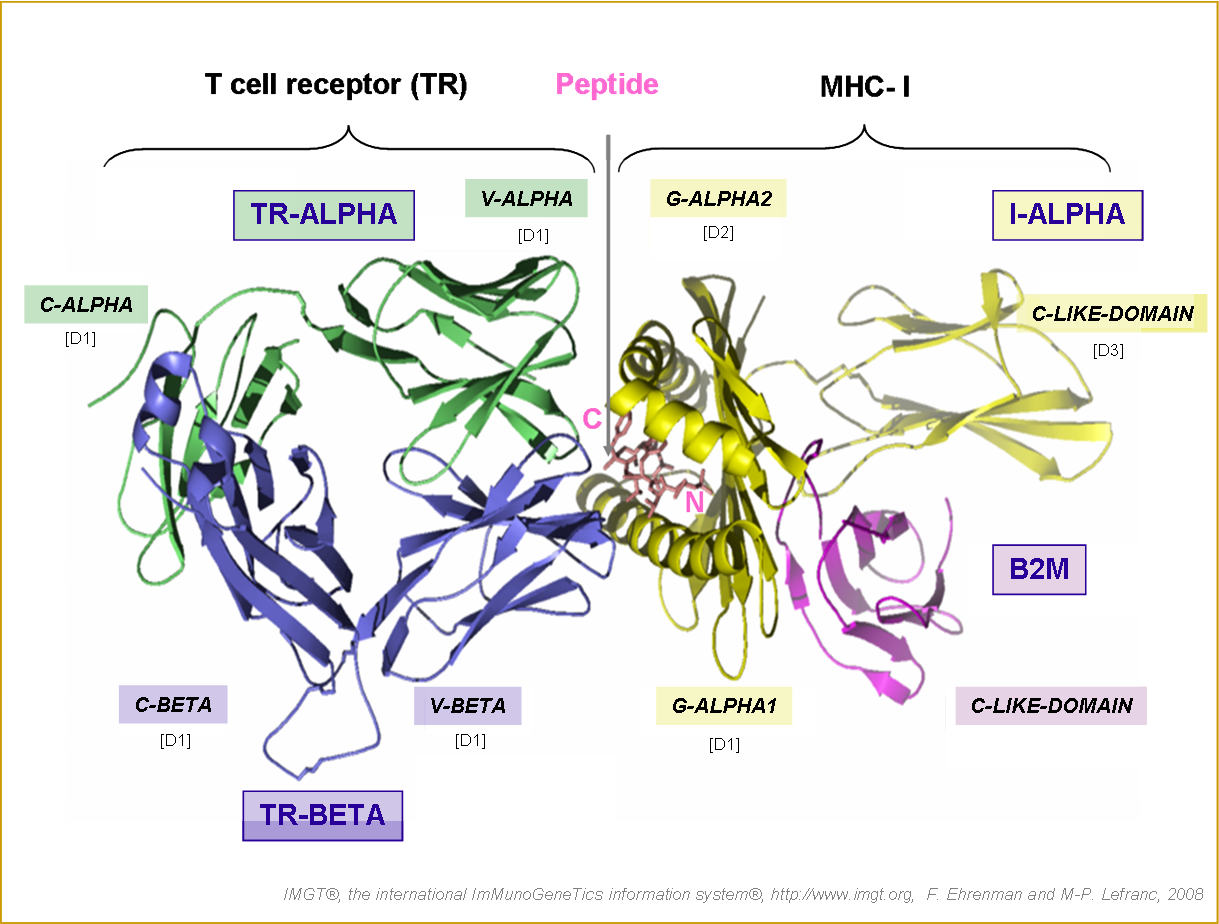 Figure 9. Complexe trimoléculaire TR/peptide/MHC-I.
Figure 9. Complexe trimoléculaire TR/peptide/MHC-I.
Le peptide (en rose) est présenté à la surface d'une cellule infectée ou d'une cellule tumorale par le MHC-I au récepteur T d'un lymphocyte T CD8+ cytotoxique. Les régions transmembranaires des chaînes du TR et du MHC-I ne sont pas présentées dans la structure 3D. IMGT/3Dstructure-DB et PDB: 1a07.
C. Structure des domaines C-LIKE des protéines MHC-I et MHC-II
1. Description du C-LIKE-DOMAIN
Les C-DOMAIN et C-LIKE-DOMAIN sont membre de la superfamille des immunoglobulines (IgSF).
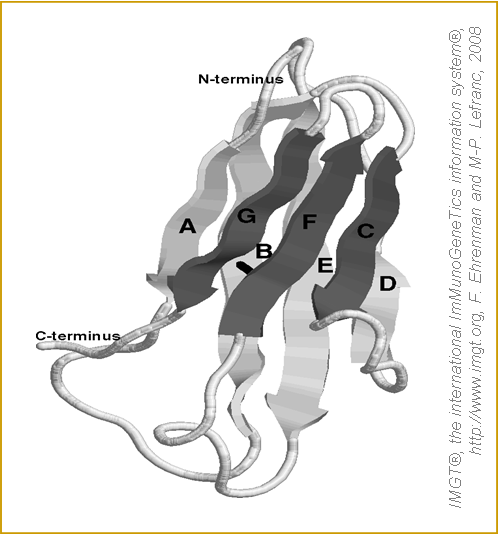
La structure du domaine C-LIKE est identique pour tous les CMH et B2M (figure 13).
Les brins A, B, D et E font partie du feuillet interne tandis que les brins C, F et G constituent le feuillet externe de la molécule CMH.
La structure du feuillet externe est très plane. Seules les trois positions 39, 40 et 41, du brin C, adoptent une conformation de type brin. Ce brin s'apparie avec le haut du brin F (positions 104 à 106).
Le bas du brin C (positions 42 à 45) n'adopte pas une structure secondaire de type brin mais forme un tour d'hélice alpha.
Le brin transversal CD (positions 745A et 45C) n'adopte pas une conformation brin.
Les brins F et G sont appariés comme représentés dans IMGT/Collier de Perles (figure14).
Figure 10. C-LIKE domain des protéines MHC-I et MHC-II. |
 |
2. Intéractions entre le C-LIKE-DOMAIN et le G-DOMAIN
Des analyses de polymorphisme des CMH montrent que les positions des classes d'hydropathie des acides aminés du C-LIKE-DOMAIN sont relativement bien conservées (figure 13).
Ainsi, les conformations des boucles BC et FG sont relativement similaires entre les différents allèles, gènes et CMH. Cette particularité peut être expliquée par le fait que ces boucles constituent des sites de contacts avec le plancher du sillon du CMH. En effet, les C-LIKE-DOMAIN se positionnent sous le feuillet bêta des G-DOMAIN (figure 9).
Par contre, on observe une variabilité structurale en ce qui conserne les brins A et D, ainsi que les boucles AB et CD.
D. Comparaison des séquences protéiques des différents gènes CMH
Il est possible de comparer les séquences protéiques CMH de différentes espèces et/ou les gènes correspondants, en utilisant l'IMGT unique numbering, les Protein displays et IMGT/Collier de Perles. En effet, une numérotation unique IMGT des G-DOMAIN et G-LIKE-DOMAIN a été mise en place (Lefranc et al. 2005a) de manière similaire à la numérotation unique IMGT des domaines IgSF (Lefranc et al. 2003 et Lefranc et al. 2005b) conformément à IMGT-ONTHOLOGY.
1. Comparaison des séquences protéiques du G-DOMAIN
- IMGT unique numbering
La numérotation unique des G-DOMAIN a été établie par une comparaison des alignements de séquences et de structures 3D des domaines G-ALPHA1 [D1], G-ALPHA2 [D2], G-ALPHA [D1], G-BETA [D1] des chaînes I-ALPHA, II-ALPHA et II-BETA.
Des positions additionnelles 7A, 14A, 54A, 61A, 61B et 92A ont été ajoutées pour constituer un standard commun de numérotation des positions des G-DOMAIN des gènes du CMH.
Ces positions correspondent à des insertions spécifiques à chaque type de G-DOMAIN afin de mettre en évidence les caractéristiques structurales locales. Ainsi, il est possible de comparer les caractéristiques de séquences, de structures et de fonctions des G-DOMAIN de différents gènes et types de CMH, mais aussi des espèces différentes.
Tableau 8. Caractéristiques des positions d'insertions des séquences MHC-I, MHC-II et MHC-I-like :
| Position d'insertion | Localisation | MHC-I | MHC-I-like | MHC-II | |||
|---|---|---|---|---|---|---|---|
| G-ALPHA1 | G-ALPHA2 | G-ALPHA1-like | G-ALPHA2-like | G-ALPHA | G-BETA | ||
| 7A | Brin A | Gap | Gap | Gap | Gap | Présence d'un AA dans la moitier des séquences* | Gap |
| 14A | Boucle AB | Gap | Gap | Gap | Gap | Gap | Présence d'un gap sauf pour Oncmyk-DAB et Oncmyk-DBB |
| 54A | Hélice | Présence d'un AA dans les MHC-I de poissons (b) | Gap | Présence d'un AA dans la majorité des MHC-like | Gap | Gap | Gap |
| 61A | Hélice | Gap | Présence d'un AA dans toutes les séquences | Gap | Présence d'un AA dans toutes les séquences | Gap dans la majorité des cas (présence d'un AA dans HLA-DMA H2-DMA) | Présence d'un AA dans toutes les séquences sauf H2-AA |
| 61B | Hélice | Gap | Gap | Gap | Présence d'un AA dans la moitier des séquences | Gap dans la majorité des cas (présence d'un AA dans HLA-DMA H2-DMA) | Présence d'un AA dans toutes les séquences sauf H2-AA |
| 72A | Hélice | Gap | Présence d'un AA dans toutes les séquences | Gap | Présence d'un AA dans toutes les séquences | Gap | Présence d'un AA dans toutes les séquences |
| 92A | Hélice | Gap | Gap | Gap | Gap | Gap dans la majorité des cas (présence d'un AA dans HLA-DMA H2-DMA) | Gap |
Cette numérotation est unique pour tous les G-DOMAIN, quelque soit le type de récepteur, le type de chaîne et l'espèce (Lefranc et al. 2005a). Elle présente ainsi un standard commun qui permet pour la première fois de comparer les domaines G-ALPHA1 avec les domaines G-ALPHA2 des MHC-I ; les domaines G-ALPHA1 (MHC-I) avec les domaines G-ALPHA (MHC-II) et les domaines G-ALPHA2 (MHC-I) avec les domaines G-BETA (MHC-II).
- Protein display
Les caractéristiques de structure des G-DOMAIN correspondant aux positions de début et de fin pour les brins et l'hélice sont reportées dans IMGT/Protein displays (Lefranc et al. 2005a). Ainsi, pour chaque feuillet d'un domain de type G:
- le brin A est défini par les positions 1 à 14,
- le brin B par les positions 18 à 28,
- le brin C par les positions 31 à 38,
- le brin D par les positions 42 à 49
- et l'hélice par les positions 50 à 92A.
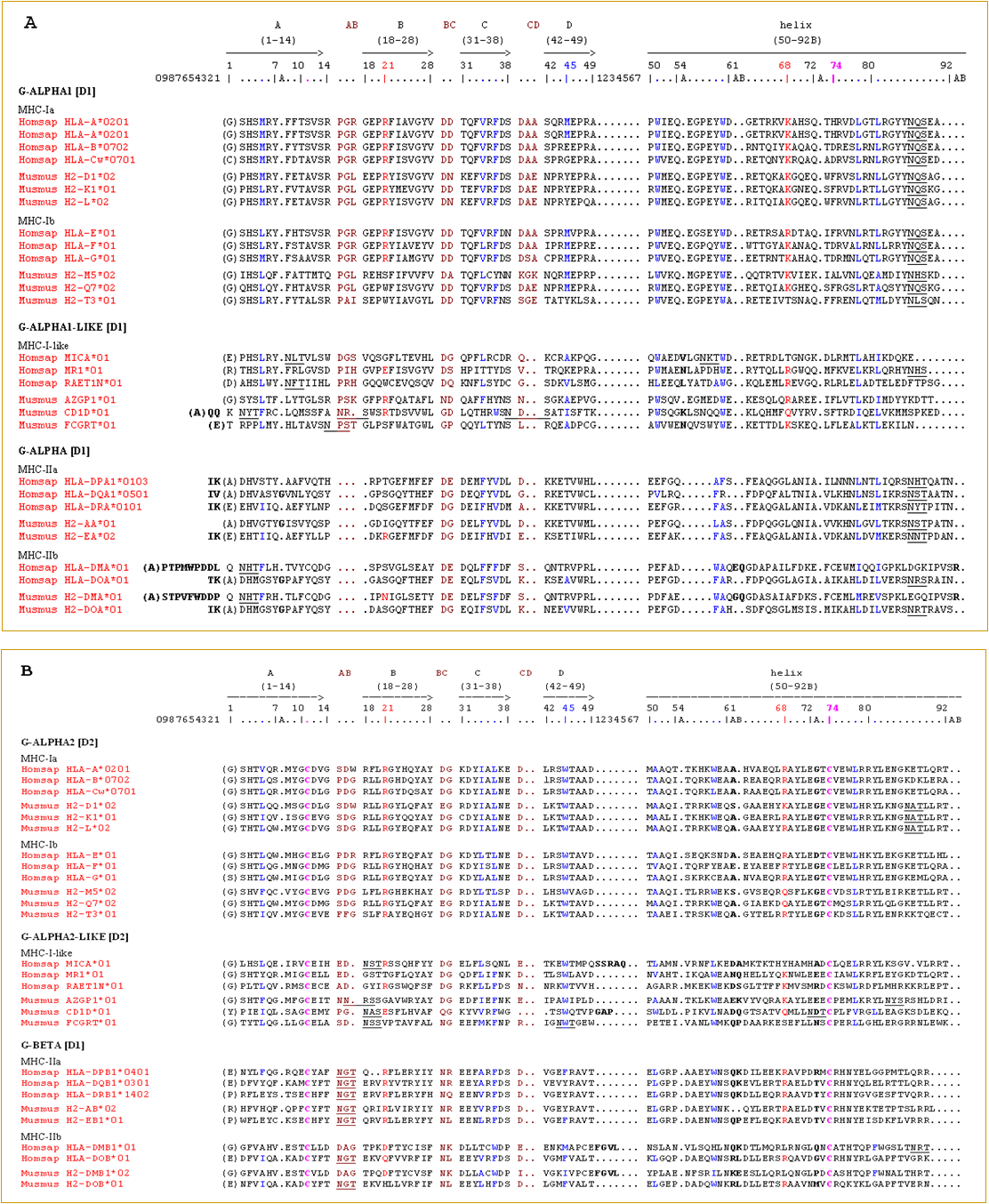 Figure 11. IMGT/Protein displays de G-DOMAIN (MHC-I et MHC-II) et G-LIKE-DOMAIN (MHC-I-like) (Lefranc et al. 2005a).
Figure 11. IMGT/Protein displays de G-DOMAIN (MHC-I et MHC-II) et G-LIKE-DOMAIN (MHC-I-like) (Lefranc et al. 2005a).
Le Protein display utilise la numérotation unique de l'IMGT basée sur les concepts de numérotation d'IMGT-ONTOLOGY. (A) Domaines G-ALPHA1 [D1] de MHC-I (MHC-Ia et MHC-Ib), G-ALPHA1-LIKE [D1] de MHC-I-like et G-ALPHA [D1] de MHC-II (MHC-IIa et MHC-IIb). (B) Domaines G-ALPHA2 [D2] de MHC-I (MHC-Ia et MHC-Ib), G-ALPHA2-LIKE [D2] de MHC-I-like et G-BETA [D1] de MHC-II (MHC-IIa et MHC-IIb). [D1], [D2] et [D3] indiquent les domaines extracellulaires des chaînes protéiques numérotés à partir de l'extrémité N-terminale des chaînes. Les points correspondent aux gaps déterminés à partir de la numérotation unique d'IMGT. Les acides aminés entre parenthèses résultent de l'épissage avec un précédent exon. Les cystéines (en magenta) correspondent aux ponts disulfures entre les cystéines C14 et C74. Les acides aminés soulignés correspondent aux sites potentiels de glycosylation. Les positions 7A, 14A, 54A, 61A, 61B, 72A et 92A indiquent des insertions dues à des variabilités ponctuelles et spécifiques de gènes ou d'espèces. EMBL/GenBank/DDBJ accession numbers: HLA-A*0201 (K02883), HLA-B*0702 (AJ292075), HLA-C *0701 (Y18533), H2-D1*02 (M18523), MH1-K1*01 (V00746), H2-L*02 (L00127), HLA-E*01 (AF523277), HLA-F*01 (X17093), HLA-G*01 (J03027), H2-M5*02 (L14279), H2-Q7*02 (X03210), H2-T3*01 (M11742), MICA*01 (U56940), MR1*01 (AL356267), RAET1N*01 (AL355497), AZGP1*01 (AF281658), CD1D1*01 (X13170), FCGRT*01 (D37872), HLA-DPA1*0103 (X03100), HLA-DPB1*0401 (M23907), HLA-DQA1*0501 (Z84489), HLA-DQB1*0301 (M25325), HLA-DRA*0101 (J00204), HLA-DRB1*1402 (AJ297583), H2-AA*02 (AY740451), H2-AB*02 (AY740477), H2-EA*02 (K00971), H2-EB1*01 (AF050157), HLA-DMA*01 (X62744), HLA-DMB*01 (X76776), HLA-DOA*01 (X02882), HLA-DOB*01 (X87344), H2-DMA*01 (AF100956), H2-DMB1*02 (U35323), H2-DOA*01 (AK020594), H2-DOB*01 (AK053233).
- IMGT/Collier de Perles
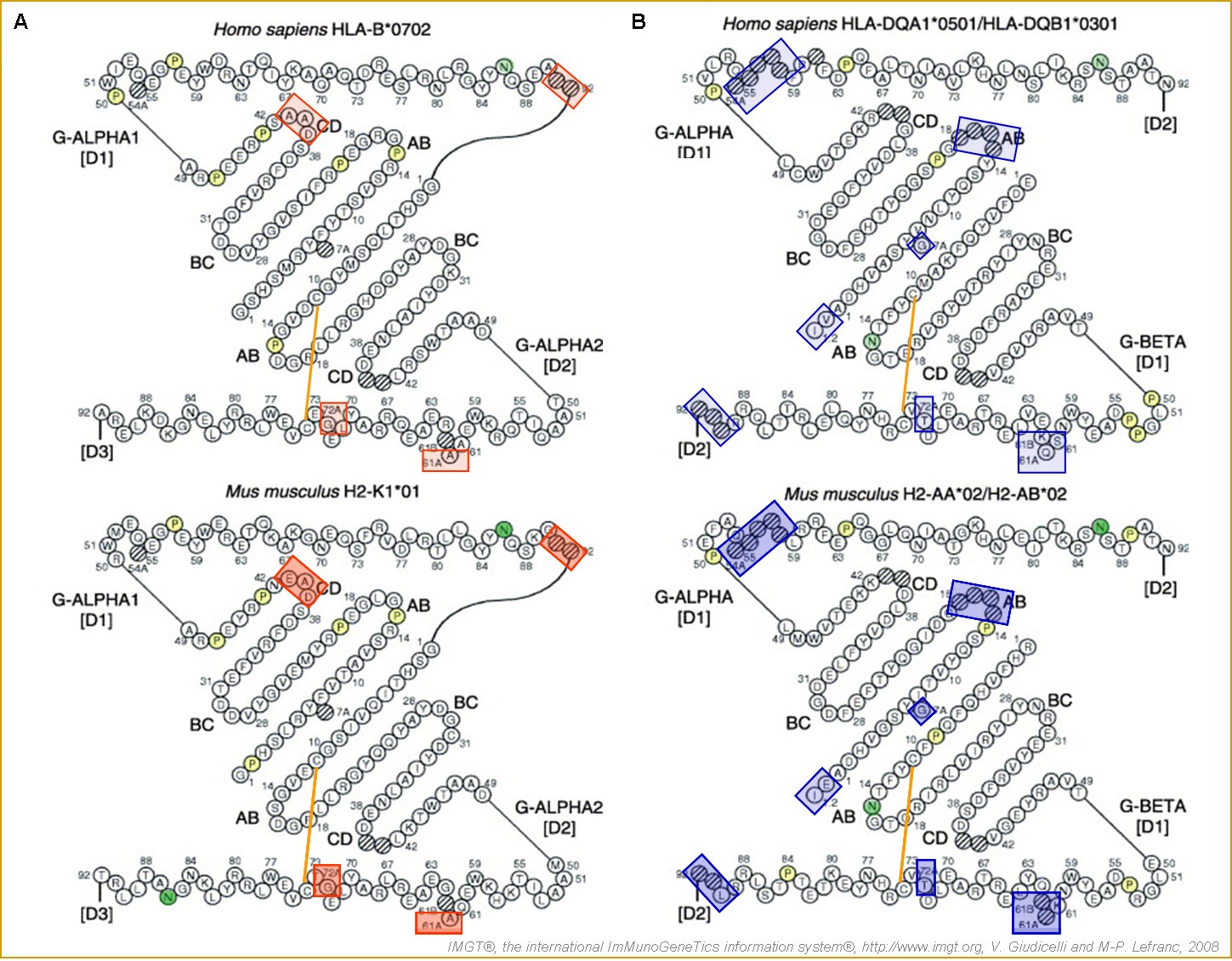 Figure 12. IMGT/Collier de Perles des G-DOMAIN de MHC-I et MHC-II (Lefranc et al. 2005a, Kaas et al. 2007) de l'homme (Homo sapiens) et de la souris (Mus musculus).
Figure 12. IMGT/Collier de Perles des G-DOMAIN de MHC-I et MHC-II (Lefranc et al. 2005a, Kaas et al. 2007) de l'homme (Homo sapiens) et de la souris (Mus musculus).
(A) Domaine G-ALPHA1 [D1] et G-ALPHA2 [D2] des protéines MHC-I HLA-B*0702 de l'homme et MH1-K1*01 de la souris. (B) Domaine G-ALPHA [D1] et G-BETA [D1] des protéines MHC-II HLA-DQA1*0501 et HLA-DQB1*0301 de l'homme et H2-AA*02 et H2-AB*02 de la souris. Les brins bêta A, B, C et D de chaque domaine sont représentés par des rangés de perles et les hélices alpha ont été déplacées en haut et en bas de la représentation afin de rendre visible le planché du sillon. L'extrémité N-terminale d'un peptide dans le sillon serait sur la gauche sur cette représentation. [D3] indique la position du C-LIKE-DOMAIN pour la chaîne I-ALPHA du MHC-I. [D2] indique la position du C-LIKE-DOMAIN pour les chaînes II-ALPHA et II-BETA du MHC-II. Les résidus proline sont en jaune. Les cadres rouges marquent les particularités des MHC-I et cadres bleus celles des MHC-II. Les chiffres indiquent les positions selon l'IMGT unique numbering. Les cercles hachurés correspondent à l'absence d'acides aminés selon IMGT unique numbering. EMBL/GenBank/DDBJ accession numbers: MH1-K1*01 (V00746), H2-AA*02 (AY740451), H2-AB*02 (AY740477). IMGT/3Dstructure-DB et PDB: MH1-K1*01 (1ld9_A), H2-AA*02 (1iak_A) et H2-AB*02 (1iak_B).
Tableau 9. Comparaison des tailles des brins antiparallèles, des boucles et hélice des G-DOMAIN des protéines MHC-I et MHC-II (Lefranc et al. 2005a) de l'homme et de la souris :
| MHC-I | MHC-II | |||
|---|---|---|---|---|
| G-ALPHA1 | G-ALPHA2 | G-ALPHA | G-BETA | |
| Brin A (A-STRAND) | 14 | 14 | 14-15 (7A (a, b)) | 14 |
| Boucle AB | 3 | 3 | 0 | 3 |
| Brin B (B-STRAND) | 11 | 11 | 10 | 11 |
| Boucle BC | 2 | 2 | 2 | 2 |
| Brin C (C-STRAND) | 8 | 8 | 8 | 8 |
| Boucle CD | 3 | 1 | 1 | 1 |
| Brin D (D-STRAND) | 8 | 8 | 8 | 8 (c) |
| Hélice | 41 | 45 (61A, 72A) | 39 (a) | 43 (d) (61A, 61B, 72A) |
| Taille totale des G-DOMAIN | 90 | 92 | 84-85 (a, b) | 94 (c, d) |
Si l'on compare les résultats obtenus entre la structure 3D et IMGT/Collier de Perles de la chaîne 1oga_A on constate:
- Une bonne correspondance pour les brins A, B et C, et les boucles qui les relient.
- Le brin D n'adopte pas une réelle conformation de brin (figures 6 et 12).
- La partie en hélice du domaine G-ALPHA1 se compose d'un segment comportant un tour d'hélice, d'une partie dépourvue de structure secondaire (positions 51 à 56) et d'un segment constitué d'une longue hélice alpha (positions 56 à 85).
- Les positions 86 à 90 de l'hélice G-ALPHA1 font le lien avec le domaine G-ALPHA2.
- La partie en hélice du domaine G-ALPHA2 se compose de deux segments (positions 51 à 61 et 63 à 85) repliés en hélice alpha .
- Les positions 61A et 62 de G-ALPHA2 relient les deux hélices alpha et sont les positions les plus élevées par rapport au plancher du sillon.
- Les domaines G-ALPHA2 et G-BETA sont caractérisés par un pont disulfure entre le brin A (CYS-11) et l'hélice (CYS-74); cette liaison est nécessaire au maintien de la structure du sillon (Saper et al. 1991).
2. Comparaison des séquences protéiques du C-LIKE-DOMAIN
- IMGT unique numbering
En raison de la forte conservation de la structure de l'immunoglobuline, la numérotation unique IMGT de C-DOMAIN et C-LIKE-DOMAIN est dérivée de la numérotation unique IMGT de V-DOMAIN (Lefranc et al. 2003).
Ainsi, la numérotation unique des C-DOMAIN et C-LIKE-DOMAIN a été établie par une comparaison des alignements de séquences et de structures 3D des V-DOMAIN, V-LIKE-DOMAIN, C-DOMAIN et C-LIKE-DOMAIN de séquences d'IG, TR et IgSF.
La numérotation unique IMGT permet de comparer entre eux:
- les domains C-DOMAIN, le C-LIKE-DOMAIN, le V-DOMAIN et le V-LIKE-DOMAIN (Lefranc et al. 2003);
- les récepteurs IG, TR, ou plus généralement IgSF;
- les chaînes de type lourd ou léger pour les IG; alpha, bêta, gamma ou delta pour les TR; ou les chaînes des protéines de IgSF;
- les espèces.
- Protein display
Les caractéristiques de structure des C-LIKE-DOMAIN sont reportées dans les IMGT/Protein displays (Lefranc et al. 2005a et b). Ainsi, pour chaque feuillet d'un domain de type C:
- le brin A est défini par les positions 1 à 15,
- le brin B par les positions 16 à 26,
- le brin C par les positions 39 à 45,
- le brin D par les positions 77 à 84,
- le brin E par les positions 85 à 96,
- le brin F par les positions 97 à 104
- et le brin G par les positions 118 à 128.
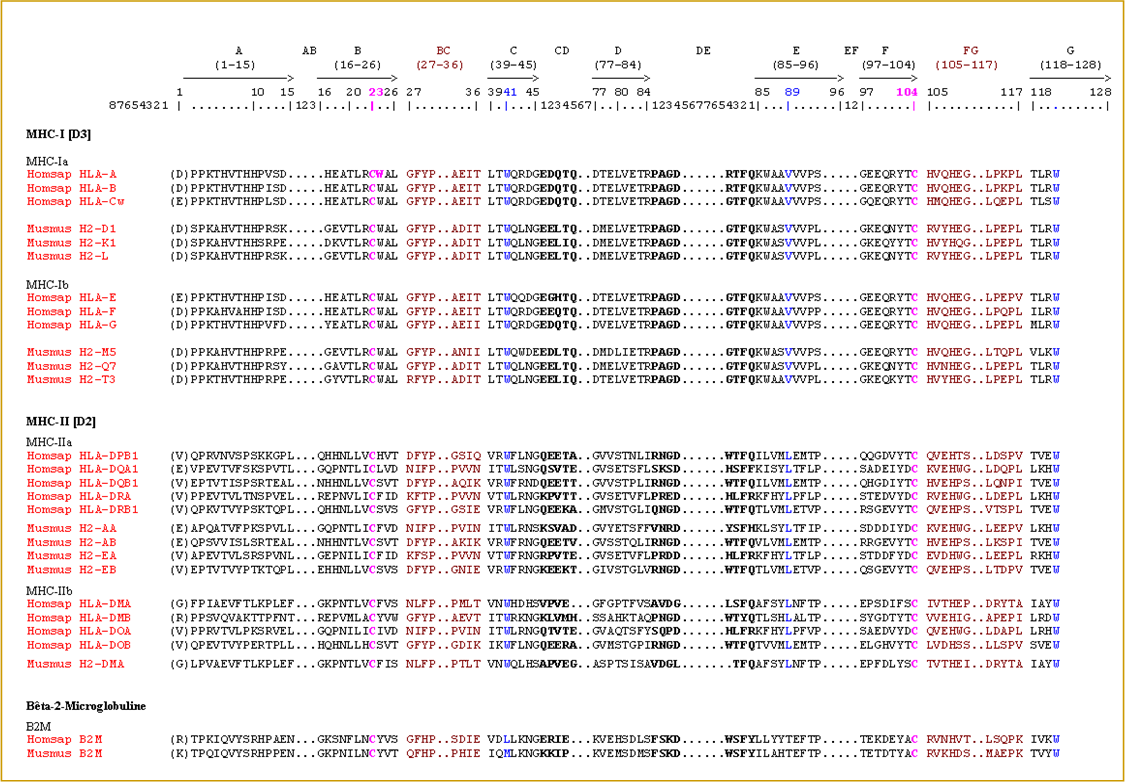 Figure 13. IMGT/Protein display des domaines de type C-LIKE des protéines MHC-I et MHC-II (Giudicelli et al. 2005, Lefranc et al. 2005a).
Le Protein display utilise la numérotation unique de l'IMGT basée sur les concepts de numérotation d'IMGT-ONTOLOGY. Les points correspondent aux gaps déterminés à partir de la numérotation unique d'IMGT. Les acides aminés entre parenthèses résultent de l'épissage avec un précédent exon. Les cystéines (en magenta) correspondent aux ponts disulfures entre les cystéines C23 et C104. IMGT/3Dstructure-DB, PDB et accession numbers: IGHG1 (J00228), IGHG4 (K01316), TRAC (1qrn_D), TRAC (1tcr_A), TRBC2 (1qrn_E), TRBC2 (1tcr_B), TRDC (1hxm_A), TRGC1 (1hxm_B), HLA-A (1a1m_A), H2-K (2vaa_A), HLA-DMA (1hdm_A), MH2-AA (1d9k_C), HLA-DMB (1hdm_B), H2-AB (1d9k_D), B2M (1a1m_B), B2M (2vaa_B).
Figure 13. IMGT/Protein display des domaines de type C-LIKE des protéines MHC-I et MHC-II (Giudicelli et al. 2005, Lefranc et al. 2005a).
Le Protein display utilise la numérotation unique de l'IMGT basée sur les concepts de numérotation d'IMGT-ONTOLOGY. Les points correspondent aux gaps déterminés à partir de la numérotation unique d'IMGT. Les acides aminés entre parenthèses résultent de l'épissage avec un précédent exon. Les cystéines (en magenta) correspondent aux ponts disulfures entre les cystéines C23 et C104. IMGT/3Dstructure-DB, PDB et accession numbers: IGHG1 (J00228), IGHG4 (K01316), TRAC (1qrn_D), TRAC (1tcr_A), TRBC2 (1qrn_E), TRBC2 (1tcr_B), TRDC (1hxm_A), TRGC1 (1hxm_B), HLA-A (1a1m_A), H2-K (2vaa_A), HLA-DMA (1hdm_A), MH2-AA (1d9k_C), HLA-DMB (1hdm_B), H2-AB (1d9k_D), B2M (1a1m_B), B2M (2vaa_B).
- IMGT/Collier de Perles

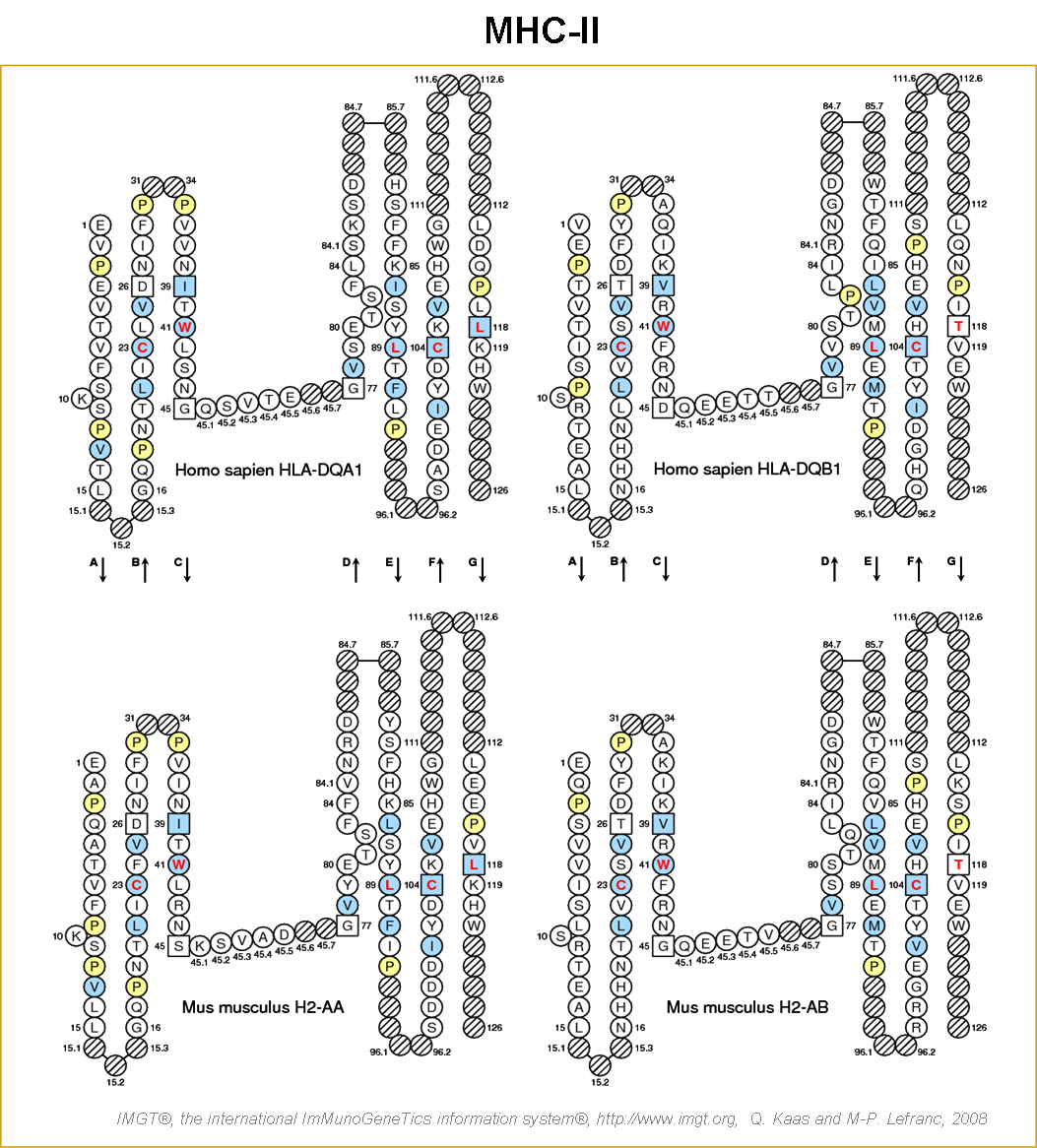
Figure 14. IMGT/Collier de Perles des domaines de type C (C-LIKE-DOMAIN) des protéines MHC-I et MHC-II (Lefranc et al. 2005b, Kaas et al. 2007). Les colliers de perles des gènes HLA-A et MH1-K1*01 sont réalisés à partir des domaines D3 de MHC-I. Les colliers de perles C-LIKE des gènes HLA-DQA1, HLA-DQB1, MH2-AA*02 et H2-AB sont réalisés à partir des domaines D2 des MHC-II. Les cercles hachurés correspondent à l'absence d'acides aminés déterminés à partir d'IMGT unique numbering. Les chiffres indiquent les positions selon IMGT unique numbering. Les positions en bleu indiquent les acides aminés hydrophobes conservés à plus de 80%. Les positions en jaune indiquent les acides aminés neutres conservés à plus de 80%. Le tryptophane en postion 41 ainsi que les cystéines en positions 23 et 104 constituent des acides aminés strictement concervés. IMGT/3Dstructure-DB et PDB : HLA-A (1a1m_A), HLA-DQA1 (NM_002122), HLA-DQB1 (FM955320), MH1-K1*01 (2vaa_A), MH2-AA (1d9k_C) et H2-AB (1d9k_D).
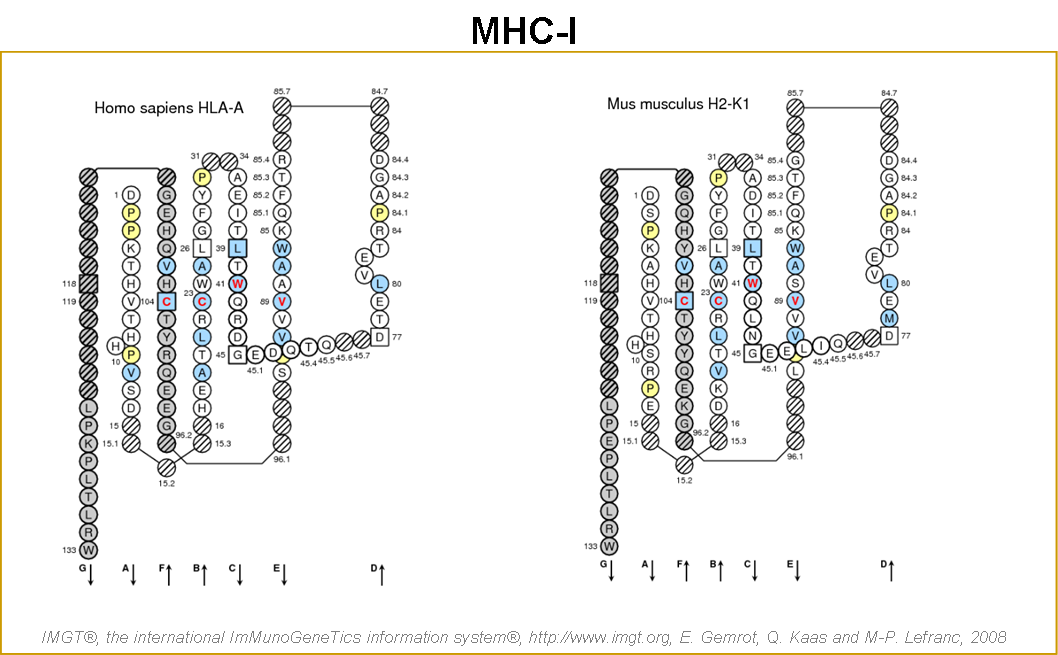
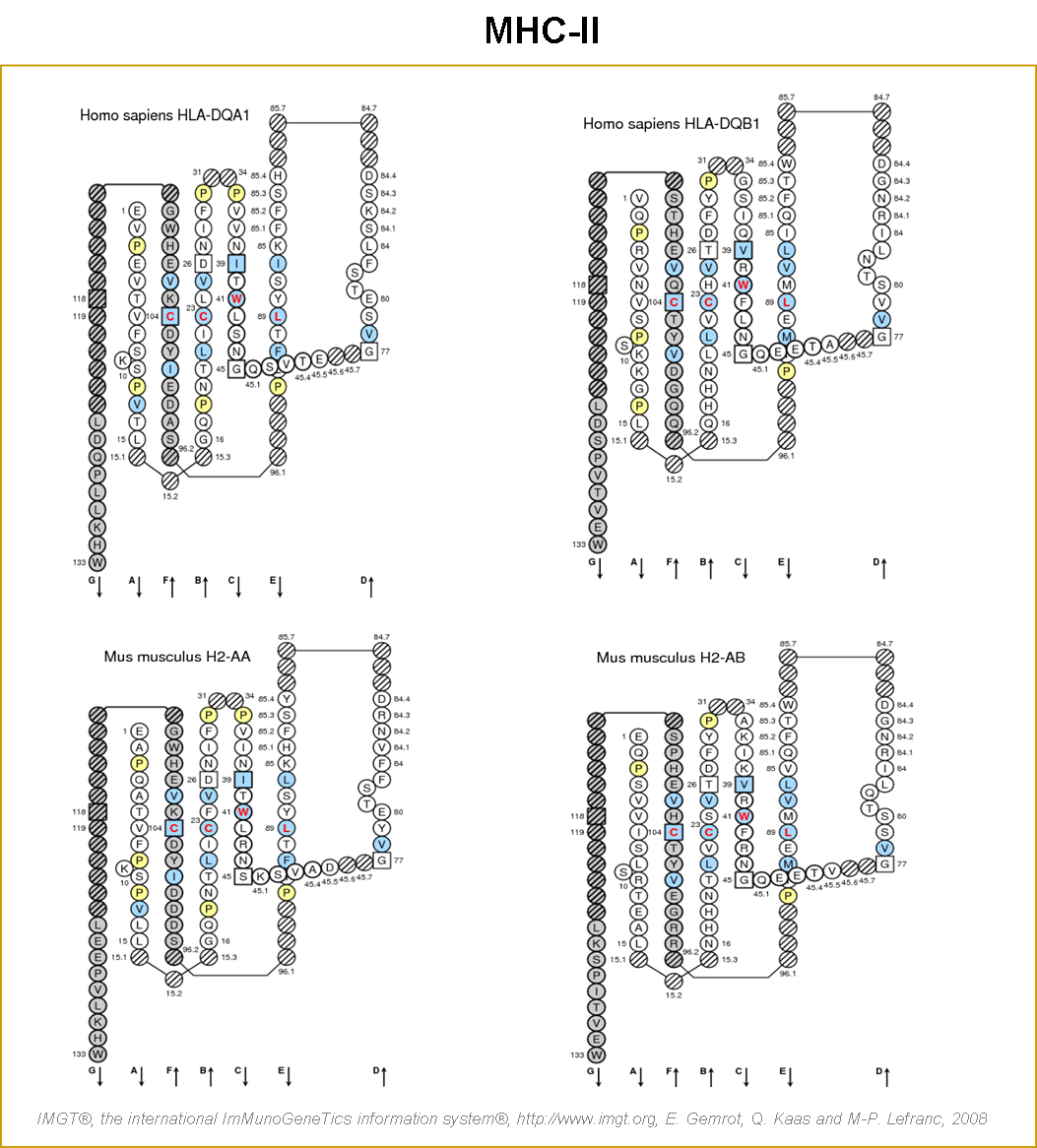
Figure 15. IMGT/Collier de Perles sur deux plans des domaines de type C (C-LIKE-DOMAIN) des protéines MHC-I et MHC-II (Lefranc et al. 2005b, Kaas et al. 2007).
Les colliers de perles des gènes HLA-A et MH1-K1*01 sont réalisés à partir des domaines D3 de MHC-I. Les colliers de perles C-LIKE des gènes HLA-DQA1, HLA-DQB1, MH2-AA*02 et H2-AB sont réalisés à partir des domaines D2 des MHC-II. Les feuillets interne du C-DOMAIN (brins A, B, E et D sont situés en arrière plan). Les feuillets externes du C-DOMAIN (brins C, F et G sont situés en avant plan). Les autres éléments du collier sont décrits dans la figure 14. IMGT/3Dstructure-DB et PDB : HLA-A (1a1m_A), HLA-DQA1 (NM_002122), HLA-DQB1 (FM955320), MH1-K1*01 (2vaa_A), MH2-AA (1d9k_C) et H2-AB (1d9k_D).
IV. Complexité du CMH
A. Généralité sur l'expression du CMH
Les relations entre le polymorphisme moléculaire, la structure, la fonction ainsi que l'expression génétique différentielle des molécules du CMH peuvent être mises en corrélation avec l'activation de la réponse immunitaire contre les pathogènes.
Contrairement aux MHC-I, les MHC-II ont une fonction régulatrice sur le système immunitaire. En effet, la densité de ce récepteur module la réponse immunitaire humorale.
Le niveau d'expression de ces récepteurs dépend du type et du degré de stress infligé aux cellules CPA. Ainsi, des facteurs physiques tels que la température (Bly et Clem 1992, Le Morvan et al. 1998 et Rodrigues et al. 1998), la présence de messages chimiques d'autres cellules ou encore la teneur en LPS (lipopolysaccharides) en cas d'invasion par des pathogènes (Knight et al. 1998 et Koppang et al. 1998), influencent le taux d'expression des molécules MHC-II au niveau transcriptionnel, traductionnel ou post traductionnel.
B. Généralité sur la diversité génétique du CMH
Pour répondre à la diversité des pathogènes et aux dérives génétiques (ex : cancer), les Gnasthostomata ont acquis une réponse immunitaire spécifique ou adaptative en diversifiant leurs molécules CMH présentatrices d'antigènes, leurs récepteurs T et leurs immunoglobulines.
Les polymorphismes génétiques et phénotypiques des CMH sont dus :
- aux divers types de CMH : MHC-Ia, MHC-Ib, MHC-IIa, MHC-IIb ;
- aux nombres d'allèles et d'haplotypes différents ;
- à l'organisation multigénique : plusieurs gènes, locus ou chromosomes ;
- à la dérive génétique : duplication, mutation et complexification ;
- au brassage génétique et recombinaisons : reproduction sexuée et crossing over ;
- à la biodiversité et à la transmission génétique : fréquences et diversités des allèles et des haplotypes en fonction des populations ;
- à la sélection naturelle et à l'adaptation génétique : résistance à des pathologies (plus un peptide aura d'affinité avec un CMH plus la réponse immunitaire sera efficace) ;
- à l'hétérozygotie et au phénotype : codominance, dominance, récessivité des allèles et haplotypes ;
- à la cellule : expression différentielle des gènes suivant le type, l'environnement et l'état de la cellule ;
- à l'espèce.
Le polymorphisme des CMH n'est pas réparti de manière égale sur toute la protéine. En effet, il est principalement localisé au niveau des sites de liaisons au peptide antigénique (G-DOMAIN) (figure 11).
De plus, le niveau de polymorphisme est fonction du type de CMH (les protéines MHC-Ib sont moins polymorphes que les protéines MHC-Ia) et au sein d'une protéine MHC-II est fonction du type de chaîne (les chaînes HLA-DRA codées par un gène unique sont moins polymorphes que les chaînes HLA-DRB codées par plusieurs gènes).
C. Comparaison des différences entre les CMH des mammifères et ceux des Teleostei
V. Fonctionnement du CMH
A. MHC-I
Les protéines MHC-I conventionnelles (MHC-Ia) sont exprimées de façon ubiquitaire à la surface des cellules nucléées. Elles présentent des peptides de 8 à 11 acides aminés majoritairement issus de la protéolyse, par le protéasome, de protéines endogènes dénaturées ou inutilisées par la cellule (Arrigo et al. 1988). Le protéasome est un complexe d'enzymes protéolytiques ATP-dépendant qui dégrade les protéines cytosoliques (après qu'elles aient été ubiquitinylées) en peptides de taille variable (3-30 AA), d'après des motifs de clivages spécifiques (prédiction in silico des sites par Kesmir et al. 2002).
1. Etapes de l'expression à la surface cellulaire d'un complexe pMHC-Ia
D'après Cresswell et al. 1999
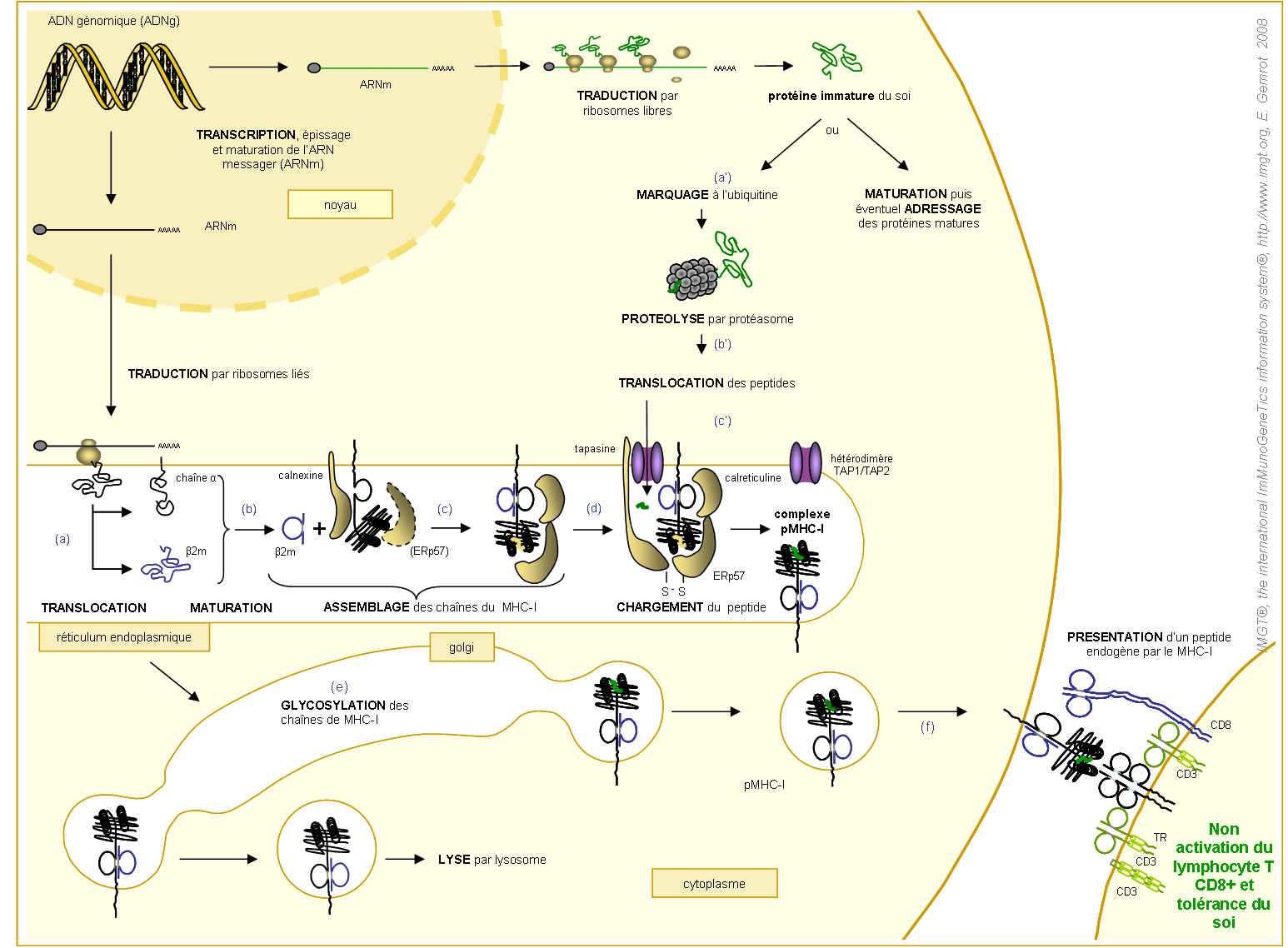 Figure 16. Etapes de l'expression à la surface cellulaire d'un complexe pMHC-Ia et présentation d'un peptide endogène par MHC-Ia au récepteur T d'un lymphocyte T CD8+ cytotoxique.
Figure 16. Etapes de l'expression à la surface cellulaire d'un complexe pMHC-Ia et présentation d'un peptide endogène par MHC-Ia au récepteur T d'un lymphocyte T CD8+ cytotoxique.
Dans le cas présent, le lymphocyte TCD8+ cytotoxiques n'est pas activé car le MHC-I présente un peptide endogène (en vert), on parle alors de « tolérance du soi ».
La voie de dégradation des peptides protéiques est indiquée par les lettres « a' » à « c' ».
La voie de biosynthèse des MHC-I et de leur chargement en peptides sont indiqués par les lettres « a » à « f ». Les différentes voies sont détaillées ci-dessous.
- Voie de dégradation des protéines endogènes (d'après Yewdell et al. 2003)
(a') Les protéines cytoplasmiques sont dégradées en permanence dans la cellule. La dégradation affecte surtout les protéines anormales en cours de synthèse (erreur traductionnelle, mauvais repliement, mutation,...) nommées plus communément DRIP (Defective Ribosomal Products) et les protéines du non soi, provenant de virus ou de bactéries à développement intracellulaire.
Les DRIP et les protéines du non soi sont marqués à l'ubiquitine. La procédure d'ubiquitination nécessite plusieurs étapes détaillées dans la figure « réactions d'ubiquitination » du module universitaire de « Biologie cellulaire », écrit par le GUIP Biologie de l'Université Bordeaux 1. Cette procedure est répètée plusieurs fois jusqu'à aboutir à la formation d'une chaîne d'au moins quatre ubiquitines qui va induire le signal de dégradation.
Les protéines polyubiquitinées sont ensuite prises en charge et dégradées en peptides au niveau du canal catalytique du protéasome ou de l'immunoprotéasome (= protéasome modifié sous l'action l'interféron).
Les protéasomes sont constitués de deux unités régulatrices PA28 (11S) et PA700 (19S) ainsi qu'une unité 20S possèdant des activités protéolytiques. La structure générale d'un protéasome est détaillées dans la figure « Composition de la particule 20S du protéasome » du module universitaire de « Biologie cellulaire », écrit par le GUIP Biologie de l'Université Bordeaux 1.
Chaque unité possède des fonctions qui lui sont propres.
- Les unités régulatrices ou « proteasome activator » limitent la dégradation non sélective des protéines en rendant accessible ou non la chambre catalytique de l'unité 20S.
L'unité régulatrice PA700, facilite le dépliage et le passage de la chaîne polypeptidique vers l'intérieur du protéasome, elle permet également le recyclage des ubiquitines (cf. figure «Fixation puis destruction par le protéasome de la beta-caténine ubiquitinée » du module universitaire de « Biologie cellulaire », écrit par le GUIP Biologie de l'Université Bordeaux 1). - La particule 20S est composée de 28 sous-unités organisées en 4 anneaux, le tout formant un cylindre. Les anneaux les plus externes (sous-unités alpha 1 à 7) n'ont pas de fonctions enzymatiques. Par contre, les 2 anneaux internes (sous-unités bêta 1 à 7) possèdent des activités protéolytiques (cf figure « Dégradation de la chaîne polypeptidique » du module universitaire de « Biologie cellulaire », écrit par le GUIP Biologie de l'Université Bordeaux 1)
- caspase (coupure après un résidu acide par la sous-unité bêta 1),
- trypsin-like (coupure après un résidu basique par la sous-unité bêta 2)
- et chymotrypsin-like (coupure après un acide aminé hydrophobe par la sous-unité bêta 5).
Il est à noter que les protéasomes sont des organites associés spatialement aux ribosomes.
(b') A la sortie du protéasome, les peptides possédant plus de 20 résidus sont clivés par la tripeptidylpeptidase (TPPII) et diverses peptidases cytosoliques. Les peptides de 7 à 20 résidus sont transportés vers la lumière du réticulum endoplasmique (RE) par le transporteur hétérodimère TAP (Transporter associated with Antigen Processing) constitué des protéines transmembranaires TAP1 et TAP2 (Kelly et al. 1992). Ces protéines sont codées par des gènes localisés dans la région du MHC-II. Elles agissent comme un filtre (Uebel et Tampé 1999), sélectionnant les peptides selon leur taille et les propriétés physico-chimiques des acides aminés à leurs extrémités. Il est à noter que le transport est actif et ATP-dépendant.
(c') Une fois dans le RE, les peptides supérieurs à 9AA sont raccourcis par l'ERAAP (endoplasmic reticulum associated amino peptidase).
- Voie de biosynthèse et de chargement en peptides des MHC-I
(a) La chaîne lourde alpha (I-ALPHA) et la bêta-2-microglobuline (B2M) sont néo-synthétisées au niveau des ribosomes liés au RE.
(b) L'assemblage de la protéine MHC-I se fait en plusieurs étapes. Dans un premier temps, la chaîne I-ALPHA est repliée et le pont disulfure C11-C74 est formé au sein du domaine G-ALPHA2 (Smith et al. 1995), grâce à la protéine chaperonne calnexine (complexée ou non à ERp57).
(c) Sous l'influence de la calnexine, la B2M établit des liaisons non covalentes avec les domaines G-ALPHA1, G-ALPHA2 et C-LIKE de la chaîne I-ALPHA.
Cet assemblage induit un changement de conformation de la chaîne I-ALPHA, contrôlé par la calréticuline (Sadasivan et al. 1996 et Mancino et al. 2002), ce qui permet la fixation d'un peptide de faible affinité (jaune) dans le sillon du CMH (sous l'action de la protéine ERAAP).
La molécule de MHC-I est stabilisée par le complexe thio-oxyréductase ERp57/calréticuline. Cet ensemble de 4 molécules forme le complexe de chargement.
(d) La thio-oxydoréductase ERp57 et la calréticuline vont permettre l'association du MHC-I à la protéine chaperone tapasine. En effet, la calréticuline interagit avec ERp57 tandis que cette dernière va oxyder la chaîne I-ALPHA du MHC-I et former des ponts disulfures avec la tapasine.
A l'heure actuelle, toutes les fonctions de la tapasine ne sont pas encore déterminées. Par ailleurs, elle interviendrait dans la régulation (Bangia et al. 1999) et la stabilisation de TAP1/TAP2. Mais aussi dans le rapprochement entre le MHC-I et le transporteur TAP1/TAP2. De ce fait, l'antigène peut se fixer plus facilement au MHC-I pour constituer un complexe stable qui ne nécessite plus la présence d'autres molécules.
Il semblerait également que la tapasine ait un rôle dans le choix d'un peptide optimal pour le MHC-I (Elliott et al. 1991). En effet, le complexe tapasine/MHC-I est en mesure d'éliminer les peptides qui ne conduisent pas à un complexe stable (Sadasivan et al. 1996 et Li et al. 1999).
(e) Les pMHC-I sont acheminés dans le compartiment de Golgi où les chaînes I-ALPHA sont glycosylées (N86 de G-ALPHA1 et chez la souris également N86 de G-ALPHA2).
(f) Enfin, le complexe pMHC-I est exprimé à la surface cellulaire.
- Présentation des antigènes à la surface des cellules
Parmi l'ensemble des peptides issus du clivage aléatoire des protéines cellulaires endogènes, 20% sont sélectionnés par les protéines TAP et seulement 0.5% sont présentés aux TR par les protéines du MHC-I. Les protéines MHC-I d'une même cellule sont néanmoins associées à un très grand nombre de peptides différents, qui représentent un échantillon des protéines endogènes. En cas d'infection ou de tumeur, certaines protéines sont produites et dégradées par le protéasome en grande quantité, ce qui conduit à la présentation majoritaire d'un ensemble de peptides par plusieurs milliers de protéines MHC-I. La probabilité de reconnaissance de ces complexes pMHC-I par des TR spécifiques est ainsi accrue.
Tableau 10. Exemples de pMHC-I présentant des peptides endogènes aux lymphocytes T CD8+ cytotoxiques :
| IMGT/3Dstructure DB | Gènes et allèles IMGT | Description | Ligands |
|---|---|---|---|
| 1seb | HLA-DRA*0101_HLA DRB1*0101 | MHC-II-ALPHA_BETA | Endogenous peptide model poly-ALA [Human] |
| 2rfx | HLA-B*5701 | MHC-I-ALPHA_B2M | Self peptide LSSPVTKSF [Synthetic] |
| 1b0g | HLA-A*0201 | MHC-I-ALPHA_B2M | Self peptide P1049 [Human] |
| 1hsa | HLA-B*2713 | MHC-I-ALPHA_B2M | Peptide [Human] |
| 1hsb | HLA-A*6801 | MHC-I-ALPHA_B2M | Peptide not identfied [Human] |
| 1i7t | HLA-A*0201 | MHC-I-ALPHA_B2M | Self peptide P1049, F>5V [Human] |
| 1i7u | HLA-A*0201 | MHC-I-ALPHA_B2M | Self peptide P1049, F>6V [Human] |
| 1lp9 | 12.2 | TR-ALPHA_BETA-1 | Self peptide P1049 [Human] |
| HLA-A*0201 | MHC-I-ALPHA_B2M |
- Cas particulier des cellules cancéreuses ou infectées par un pathogène
- Cas d'une cellule cancéreuse
- Cas d'une cellule infectée par une bactérie pathogène (ex : Listeria monocytogenes)
- Cas d'une cellule infectée par un virus à ADN (ex : Adenoviridae)
- Cas d'une cellule infectée par un virus à ARN (ex : VIH)
Tableau 11. Comparaison de la taille de différentes entités :
| Entités | Taille | |
|---|---|---|
| Protéines | Antigènes du CMH | 0,5-2nm |
| Anticorps | 14-20nm | |
| Virus | Adenovirus (virus ADN) | 60-90nm |
| VIH (virus ARN) | 80-120nm | |
| Bactérie | Lactococcus lactis (cocci) | 0,5-1,5µm |
| Listeria monocytogenes (bacille) | 1-4µm de long contre 0.5µm de large | |
| Cellules immunitaires |
Lymphocytes T et B | 7-10µm |
| Natural killer | 12-14µm | |
| Monocyte et cellule dendritique | 15-30µm | |
| Macrophage | 20 à 70µm | |
2. Fonctionnement des protéines MHC-Ia et MHC-Ib
Les protéines MHC-Ib suivent la même voie d'expression que les protéines du CMH conventionnelles (MHC-Ia) mais diffèrent par la nature des peptides qu'elles présentent aux TR et par les populations cellulaires respectives qui les expriment (Braud et al. 1999).
Les protéines MHC-Ia sont ubiquitaires et présentent des peptides issus de la dégradation de protéines endogènes; elles interagissent également avec différents récepteurs inhibiteurs des cellules NK appartenant aux familles multigéniques KIR (Snyder et al. 1999), LIR (Cosman et al. 1997) et NKG2 (Tormo et al. 1999). Les cellules NK sont des lymphocytes qui détectent certaines anomalies à la surface des cellules de l'organisme et les détruisent (Trinchieri 1989 et Biron 1997). Les anomalies détectées correspondent à une réduction du nombre de protéines MHC-I ou au profil inhabituel des antigènes de surface des cellules tumorales ou des cellules infectées par des virus (Ljunggren et Karre 1990).
L'interaction des protéines MHC-Ib avec les récepteurs des cellules NK induit la transduction d'un signal inhibiteur de l'activité cytotoxique de ces cellules tueuses.
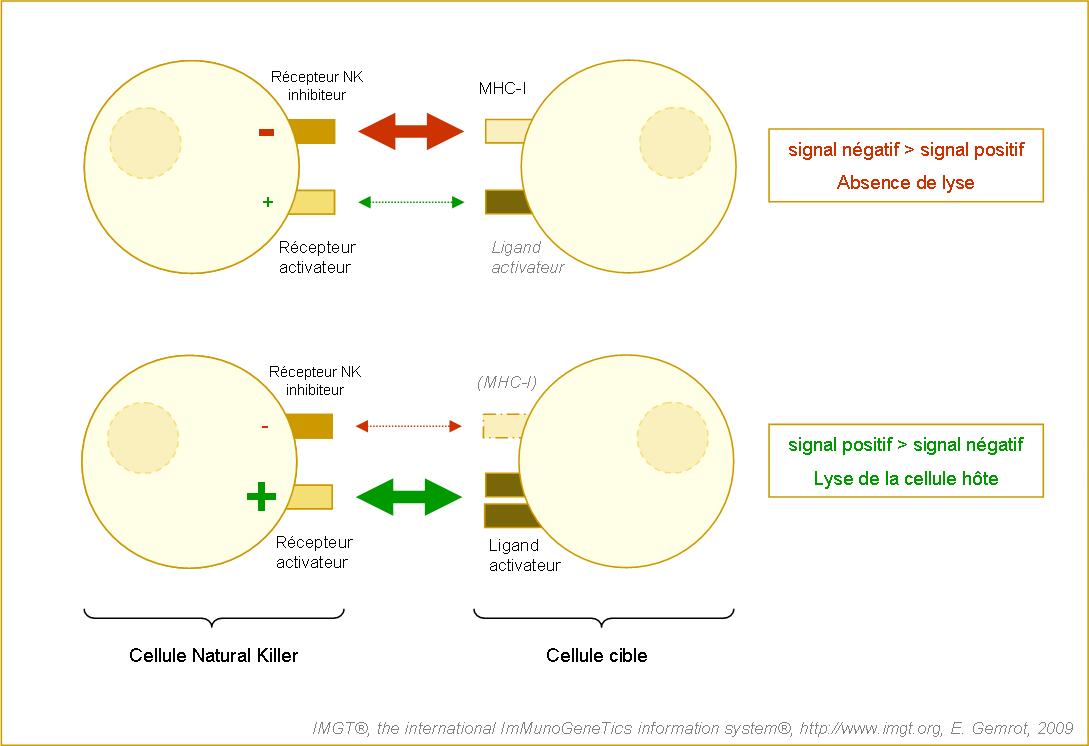 Figure 17. Mécanismes d'activation et de répression de l'activité cytotoxique des cellules NK vis-à-vis d'une cellule cible (Schleinitz et al. 2002).
Figure 17. Mécanismes d'activation et de répression de l'activité cytotoxique des cellules NK vis-à-vis d'une cellule cible (Schleinitz et al. 2002).
L'activation ou l'inhibition de la capacité cytotoxique des cellules NK résulte de l'intégration des différents signaux positifs (en vert) et négatifs (en rouge).
- Dans le cas d'une cellule cible saine et appartenant au soi, l'activité cytotoxique des cellules NK est bloquée par la transmission majoritaire de signaux négatifs. Ces signaux proviennent de la reconnaissance des molécules MHC-I, suffisament exprimées, par les récepteurs NK inhibiteurs.
- Dans le cas d'une cellule stressée ou du non soi, la capacité cytotoxique des cellules NK est activée par la transmission majoritaire de signaux positifs due à la reconnaissance spécifique de ligands surexprimés et à une diminution de l'expression de protéines du MHC-I.
Ainsi, prenons pour exemple, le cas d'une femme enceinte. La molécule HLA-G est exprimée à la limite foeto-maternelle (cytotroblaste extravillieux, où les MHC-Ia sont absents), afin de protéger le fœtus contre les cellules NK. En effet, les HLA-G solubles se lient sur les récepteurs KIR (Killer Inhibitory Receptor) qui induisent l'inhibition des fonctions lytiques (perforine, cytoxine...) des cellules NK (Martal J et al. 2002).
3. Fonctionnement de la bêta-2-microglobuline (B2M)
La B2M joue un rôle essentiel au cours du processus d'expression des protéines MHC-Ia (Arce-Gomez et al. 1978) et MHC-Ib, en particulier pour :
- la liaison du peptide (Boyd et al. 1992). La B2M est liée spécifiquement par la protéine TAP-1 (Androlewicz et al. 1994) et assure le maintien de la conformation du site de liaison au peptide (Townsend et al. 1990). Cette stabilité est essentielle pendant l'échange entre les peptides de faible et de haute affinité. La précédence de la liaison de B2M sur celle du peptide a été mise en évidence in vivo (Neefjes et al. 1993 et Ortmann et al. 1994).
- la stabilité de la structure des protéines MHC-Ib (Hill et al. 2003).
- la glycosylation de la chaîne lourde des protéines MHC-I dans l'appareil de Golgi (Sege et al. 1981).
- la reconnaissance des protéines MHC-Ib par certains récepteurs des cellules NK à la surface cellulaire : Ly49A chez Mus musculus (Tormo et al. 1999 et Michaëlsson et al. 2001), NKG2A (Yokoyama 1998) et les protéines de la famille multigénique LIR (Willcox et al. 2003) chez Homo sapiens.
Les liaisons établies entre la B2M et le domaine C-LIKE de la chaîne lourde de MHC-I ont longtemps été considérées comme seules requises pour l'interaction I-ALPHA/B2M; les expériences de mutagenèse dirigée destinées à comprendre ce qui détermine cette interaction protéine-protéine étaient donc limitées à ce domaine protéique. En 1995, Collins et al. ont mis en évidence que la délétion in vitro du domaine C-LIKE d'une chaîne I-ALPHA n'affecte ni sa structure, ni sa liaison à la B2M et au peptide. Les liaisons établies entre la B2M et les domaines G-ALPHA des protéines MHC-I sont donc considérées avec attention depuis cette expérience (Hill et al. 2003 et Duprat et al. 2006). L'échange de B2M intraspécifique et interspécifique à la surface cellulaire semble être un phénomène courant, mis en évidence in vitro (Bernabeu et al. 1984).
B. MHC-II
Les protéines MHC-II conventionnelles (MHC-IIa) présentent des peptides exogènes de 12 à 25 acides aminés, issus de la protéolyse à pH acide de protéines endocytées à partir de la surface cellulaire.
Le nombre de peptides différents susceptibles de s'associer avec les protéines MHC-IIa est plus élevé que dans le cas des protéines MHC-I, du fait de l'absence de sélection par des protéines chaperonnes, et de l'hétérogénéité des poches de liaison au niveau des domaines G-ALPHA et G-BETA. Les éléments critiques pour cet assemblage sont :
- l'efficacité de la protéolyse de la chaîne invariante Li,
- l'avidité de la protéine MHC-IIa pour Li et pour les peptides,
- l'expression des protéines MHC-IIb,
- la capacité des peptides à résister aux protéases multicatalytiques dans les endosomes,
- et la stabilité structurale qui résulte de cet assemblage.
Voies de biosynthèse et de chargement en peptides des MHC-II :
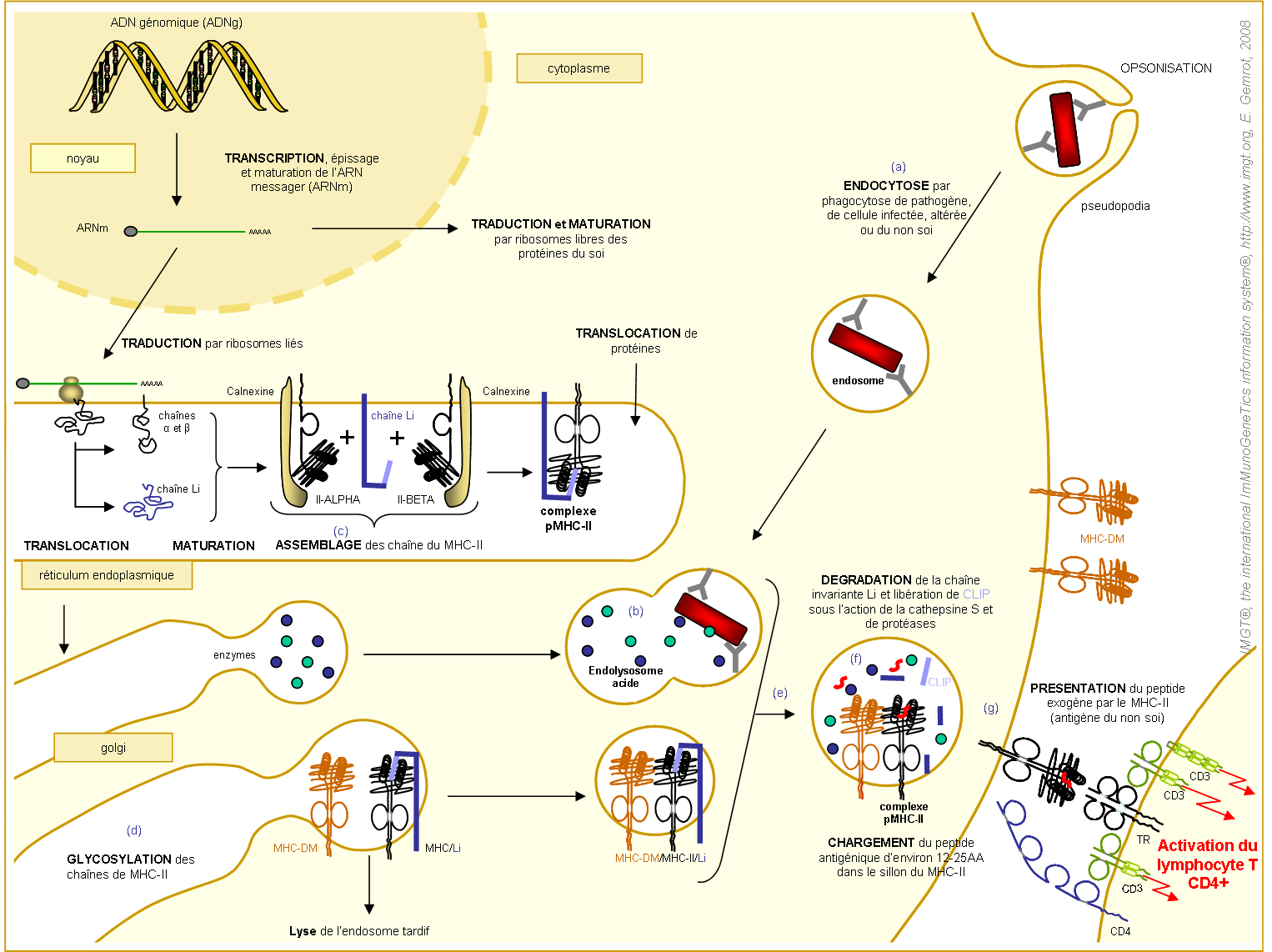 Figure 18. Etapes de l'expression à la surface cellulaire d'un complexe pMHC-IIa et présentation d'un peptide exogène par MHC-IIa au récepteur T d'un lymphocyte T CD4+.
Figure 18. Etapes de l'expression à la surface cellulaire d'un complexe pMHC-IIa et présentation d'un peptide exogène par MHC-IIa au récepteur T d'un lymphocyte T CD4+.
Dans le cas présent, le lymphocyte TCD4+ est activé car le MHC-II présente un peptide exogène (en rouge) issu de la dégradation d'un pathogène ou d'une cellule altérée ou du non soi.
La voie de dégradation des peptides protéiques exogène nécessite une lyse dans un endozome acide.
La voie de biosynthèse des MHC-II et de leur chargement en peptides sont indiqués par lettres « a » à « f ». Les différentes voies sont détaillées ci-dessous.
L'expression à la surface cellulaire d'un complexe pMHC-IIa nécessite six étapes, au sein desquelles les protéines MHC-II non conventionnelles (MHC-IIb) jouent un rôle essentiel (Chapman 1998) :
(a) Les protéines extracellulaires de bactéries, de cellules infectées, altérées ou étrangères sont dans un premier temps endocytées.
(b) Puis, elles sont dégradées par des enzymes dépendantes ou non de l'O2 (Burst oxydatif) au niveau des vésicules d'endocytose fusionnées ou non avec des lysosomes; ce qui génère des peptides de tailles diverses. Le pH de ces endolysosomes formés est d'environ 4,5.
(c) Dans le réticulum endoplasmique, la chaîne invariante Li et les protéines MHC-IIa s'associent avec l'aide de protéines chaperonnes, telles que la calnexine et la chaîne Li.
La chaîne invariante Li a trois fonctions :
- elle stabilise la molécule de MHC-II dépourvue de peptide,
- empêche la fixation de peptides endogènes, destinés au MHC-I, durant la traversée du RE
- et permet l'adressage de l'ensemble du complexe Li/MHC-II vers des endosomes tardifs.
Il est à remarquer que l'affinité de la chaîne Li varie selon la classe et l'allèle de la protéine MHC-IIa (Villadangos et Ploegh 2000).
(d) Les chaînes II-ALPHA et II-BETA des protéines MHC-IIa sont glycosylées dans l'appareil de golgi et l'adressage du complexe Li/MHC-II dans le compartiment cellulaire des MHC-II se fait ensuite par le peptide signal de Li (Bakke et Dobberstein 1990).
(e) Les vésicules du compartiment cellulaire des MHC-II fusionnent avec des endosomes ou des endolysosomes. Sous l'influence du pH acide et des protéases de ces compartiments (cathepsines B, D, L et S), la chaîne Li se dissocie et laisse le peptide CLIP (class II-associated invariant chain peptide) dans le sillon de la protéine MHC-IIa (Cresswell 1996). CLIP correspond aux résidus 81-104 de la chaîne Li et est issu de sa protéolyse (Zhu et al. 2003).
(f) CLIP est échangé avec un peptide présent dans l'endosome ou le lysosome. La protéine chaperonne MHC-IIb MHC-DM reconnaît spécifiquement les protéines MHC-IIa et catalyse cet échange (Mosyak et al. 1998) en réalisant dans un premier temps une association transitoire entre la molécule MHC-DM, le MHC-II et CLIP. Cette interaction va changer la conformation de la protéine MHC-II, libérant ainsi le sillon où se nichera le peptide. Une fois que l'un des peptides est fixé de manière stable au MHC-II, MHC-DM est recyclée. Puis, la protéine MHC-IIb MHC-DO, exprimée dans les lymphocytes B, inhibe l'action de MHC-DM. Cette inhibition est dépendante du pH et permet d'accroître la présentation de peptides issus des antigènes internalisés suite à leur reconnaissance par les Ig membranaires (Liljedahl et al. 1998).
(g) Transport des complexes pMHC-IIa jusqu'à la surface cellulaire. Une partie des protéines MHC-II membranaires sont ensuite internalisées dans les endosomes et recyclées à la surface.
VI. Bibliographie
Androlewicz, M.J., Ortmann, B., van Endert, P.M., Spies, T., Cresswell, P., Characteristics of peptide and major histocompatibility complex class I/beta2-microglobulin binding to the transporters associated with antigen processing (TAP1 and TAP2), Proc. Natl. Acad. Sci. USA., 91,2716-12720 (1994).
Arce-Gomez, B., Jones, E.A., Barnstable, C.J., Solomon, E., Bodmer, W.F., The genetic control of HLA-A and B antigens in somatic cell hybrids: requirement for beta2 microglobulin, Tissue Antigens, 11, 96-112 (1978).
Arrigo, A.P., Tanaka, K., Goldberg, A.L., Welch, W.J., Identity of the 19S 'prosome' particle with the large multifunctional protease complex of mammalian cells (the proteasome), Nature, 331, 192-194 (1988).
Bakke, O., Dobberstein, B., MHC class II-associated invariant chain contains a sorting signal for endosomal compartments, Cell, 63, 707-716 (1990).
Bangia, N., Lehner, P.J., Hughes, E.A., Surman, M., Cresswell, P., The N-terminal region of tapasin is required to stabilize the MHC class I loading complex, Eur J Immunol., 29(6), 1858-70 (1999).
Berman, H.M., Westbrook, J., Feng, Z., Gilliland, G., Bhat, T.N., Weissig, H., The Protein Data Bank. Nucl. Acids Res., 28, 235-42 (2000).
Bernabeu, C., van de Rijn, M., Lerch, P and Terhost, C., beta2microglobulin from serum associates with class I antigens on the surface of cultured cells, Nature, 308, 642-645 (1984).
Biron, C.A., Activation and function of natural killer cell responses during viral infections, Curr. Opin. Immunol., 9, 24-34 (1997).
Bjorkman, P.J.,Strominger J.L. ; Wiley, D.C., Crystallization and X-ray diffraction studies on the histocompatibility antigens HLA-A2 and HLA-A28 from human cell membranes, Journal of molecular biology, 186(1), 205-210 (1985).
Bjorkman, P.J., Parham, P., Structure, Function, and Diversity of Class I Major Histocompatibility Complex Molecules, Annual Review of Biochemistry, 59, 253-288 (1990).
Bly, J.E., Clem, L., Temperature and teleost immune functions, Fish Shellfish Immunol., 2, 159-171 (1992).
Boyd, L.F., Kozlowski, S., Margulies, D.H., Solution binding of an antigenic peptide to a major histocompatibility complex class I molecule and the role of B2-microglobulin, Proc. Natl. Acad. Sci. USA, 89, 2242-2246 (1992).
Braud, V.M., Allan, D.S.J., McMichael, A.J., Functions of non classical MHC and non-MHC-encoded class I molecules, Curr. Opin. Immunol., 11, 100-108 (1999).
Brown, J.H., Jardetzky, T.S., Gorga, J.C., Stern, L.J., Urban, R.G., Strominger, J.L., Wiley, D.C., Three-dimensional structure of the human class II histocompatibility antigen HLA-DR1, Nature, 364, 33-39 (1993).
Chapman, H.A., Endosomal proteolysis and MHC-II function, Curr. Opin. Immunol., 10, 93-102 (1998).
Clark, M.S., Shaw, L., Kelly, A., Snell, P., Elgar, G., Characterization of the MHC class I region of the Japanese pufferfish (Fugu rubripes), Immunogenetics, 52, 174-185 (2001).
Collins, E.J., Garboczi, D.N., Karpusas, M.N., Wiley, D.C., The three-dimensional structure of a class I major histocompatibility complexe molecule missing the alpha 3 domain of the heavy chain, Proc. Natl. Acad. Sci. USA, 92, 1218-1221 (1995).
Cosman, D., Fanger, N., Borges, L., Kubin, M., Chin, W., Peterson, L., Hsu, M.-L., A novel immunoglobulin superfamily receptor for cellular and viral MHC class I molecules, Immunity, 7, 273-282 (1997).
Cresswell, P., Invariant chain structure and MHC class II function, Cell, 84, 505-507 (1996).
Cresswell, P., Bangia, N., Dick, T. and Diedrich, G., The nature of the MHC class I peptide loading complex, Immunol. Rev., 172, 21-28 (1999).
Duprat, E., Lefranc, M.P., Gascuel, O., A simple method to predict protein binding from aligned sequences - application to MHC superfamily and beta2-microglobulin, Bioinformatics, 22, 453-459 (2006).
Elliott, T., Cerundolo, V., Elvin, J. and Townsend, A., Peptide-induced conformational change of the class I heavy chain, Nature, 351, 402-406 (1991).
Elliott T., The 'chop-and-change' of MHC class I assembly. Nature Immunol., 7, 7-9 (2006).
Falk, K., Rötzschke, O., Stevanovic, S., Jung, G., Rammensee, H.G. , Allele-specific motifs revealed by sequencing of self-peptides eluted from MHC molecules, Nature, 651, 290-296 (1991).
Feder, J.N., Penny, D.M, Irrinki, A., Lee, V.K., Lebron, J.A., Watson, N., and al., The hemochromatosis gene product complexes with the transferrin receptor and lowers its affinity for ligand binding, Proc Natl Acad Sci USA, 95, 1472-1477 (1998).
Frigoul, A., Lefranc, M.P., MICA: standardized IMGT allele nomenclature, polymorphisms and diseases, In: Recent Research Developments in Human Genetics (Pandalai S.G., ed.), Research Signpost, Trivandrum, Kerala, India, 3, 95-145 (2005).
Giudicelli, V., Chaume, D., Lefranc, M-P., IMGT/GENE-DB: a comprehensive database for human and mouse immunoglobulin and T cell receptor genes, Nucl Acids Res, 33,D256-D261 (2005).
Gopalakrishnan, B., Roques, B. P., Do antigenic peptides have a unique sense of direction inside the MHC binding groove ? A molecular modelling study, FEBS letters, 303(2-3), 224-228 (1992).
Hill, D.M., Kasliwal, T., Schwarz, E., Hebert, A.M., Chen, T., Gubina, E., Zhang, L., Kozlowski, S., A dominant negative mutant B2-microglobulin blocks the extracellular folding of a major histocompatibility complex class I heavy chain, J. Biol. Chem., 278, 5630-5638 (2003).
Holmes, M.A., Li, P., Petersdorf, E.W., Strong, R.K., Structural Studies of Allelic Diversity of the MHC Class I Homolog MIC-B, a Stress-Inducible Ligand for the Activating Immunoreceptor NKG2D, The J.Immunol., 169, 1395-1400 (2002).
Kaas, Q., Ruiz, M., Lefranc, M.-P., IMGT/3Dstructure-DB and IMGT/StructuralQuery, a database and a tool for immunoglobulin, T cell receptor and MHC structural data, Nucleic Acids Res, 32, D208-D10 (2004).
Kaas, Q., Lefranc, M.-P., IMGT Colliers de Perles: standardized sequence-structure representations of the IgSF and MhcSF superfamily domains, Current Bioinformatics, 2, 21-30 (2007).
Karlsson, L., DM and DO shape the repertoire of peptide-MHC-class-II complexes, Curr Opin Immunol., 17, 65-70 (2005).
Kelly, A., Powis, S.H., Kerr, L.A., Mockridge, I., Elliott, T., Bastin, J., Uchanska-Ziegler, B., Ziegler, A., Trowsdale, J., Townsend, A., Assembly and function of the two ABC transporter proteins encoded in the human major histocompatibility complex, Nature, 355, 641-644 (1992).
Kesmir, C., Nussbaum, A.K., Schild, H., Detours V. and Brunak S., Prediction of proteasome cleavage motifs by neural networks, Protein Eng., 15, 287-296 (2002).
Kjer-Nielsen, C., Clements, A., Purcell, A., Brooks, J., Whisstock, S., Burrows, J., McCluskey, J., Rossjohn, A., Structural Basis for the Selection of Dominant αβ T Cell Receptors in Antiviral Immunity, Immunity, 8, 53-64 (2003).
Knight, J., Stet, R.J.M., Secombes, C.J., Modulation of MHC class II expression in rainbow trout Oncorhynchus mykiss macrophages by TNFa and LPS, Fish Shellfish Immunol., 8, 545-553 (1998).
Koppang, E.O., Lundin, M., Press, C. McL., Ronningen, K., Lie, O., Differing levels of Mhc class II b chain expression in a range of tissues from vaccinated and non-vaccinated Atlantic salmon (Salmo salar L.), Fish Shellfish Immunol., 8, 183-196 (1998).
Kulski, J.K., Shiina, T., Anzai, T., Kohara, S., Inoko, H., Comparative genomic analysis of the MHC: the evolution of class I duplication blocks, diversity and complexity from shark to man. Immunol., Rev 190, 95-122 (2002).
Lefranc, M.P., Pommié, C., Ruiz, M., Giudicelli, V., Foulquier, E., Truong, L., Thouvenin-Contet, V., Lefranc, G., IMGT unique numbering for immunoglobulin and T cell receptor variable domains and Ig superfamily V-like domains, Dev. Comp. Immunol., 27(1), 55-77 (2003).
Lefranc, M.-P., Duprat, E., Kaas, Q., Tranne, M., Thiriot, A. and Lefranc, G., IMGT unique numbering for MHC groove G-DOMAIN and MHC superfamily (MhcSF) G-LIKE-DOMAIN, Dev. Comp. Immunol., 29, 917-938 (2005a).
Lefranc, M.-P., Pommié, C., Kaas, Q., Duprat, E., Bosc, N., Guiraudou, D., Jean, C., Ruiz, M., Da Piedade, I., Rouard, M., Foulquier, E., Thouvenin, V., Lefranc, G., IMGT unique numbering for immunoglobulin and T cell receptor constant domains and Ig superfamily C-like domains, Dev. Comp. Immunol., 29, 185-203 (2005b).
Le Morvan, C., Troutaud, D., Deschaux, P., Differential effects of temperature on specific and nonspecific immune defences in fish, J. Exp. Biol., 201, 165-168 (1998).
Lepin, E.J., Bastin, J.M., Allan, D.S., Roncador, G., Braud, V.M., Mason, D.Y., van der Merwe, P.A., McMichael, A.J., Bell, J.I., Powis, S.H., O'Callaghan, C.A., Functional characterization of HLA-F and binding of HLA-F tetramers to ILT2 and ILT4 receptors, Eur J Immunol., 30(12), 3552-61 (2000).
Li, S., Paulsson, K.M., Sjögren, H.-O., Wang, P., Peptide-bound major histocompatibility complex class I molecules associate with tapasin before dissociation from transporter associated with antigen processing, J. Biol. Chem., 274, 8649-8654 (1999).
Li, P., McDermott, G., Strong, R.K., Crystal structures of RAE-1beta and its complex with the activating immunoreceptor NKG2D, Immunity, 16(1), 77-86 (2002).
Liljedahl, M., Kuwana, T., Fung-Leung, W.P, Jackson, M.R, Peterson, P.A, Karlsson, L., HLA-DO is a lysosomal resident which requires association with HLA-DM for efficient intracellular transport, EMBO J 15, 4817-4824 (1996).
Liljedahl, M., Winqvist, O., Surh, C.D., Wong, P., Ngo,, K., Teyton, L., Peterson, P.A., Brunmark, A., Rudensky, A.Y., Fung-Leung, W.P., Karlsson, L., Altered antigen presentation in mice lacking H2-O, Immunity, 8, 233-243 (1998).
Ljunggren, H.G., Karre, K., In search of the 'missing self': MHC molecules and NK cell recognition, Immunol., 11, 237-244 (1990).
Lukacs, M.F., Harstad, H., Grimholt, U., Beetz-Sargent, M., Cooper, G.A., Reid, L., Bakke, H.G., Phillips, R.B., Miller, K.M., Davidson, W.S., Koop, B.F., Genomic organization of duplicated major histocompatibility complex class I regions in Atlantic salmon (Salmo salar), BMC Genomics, 8, 251 (2007).
Mancino, L., Rivzi, S.M., Lapinski, P.E., Raghavan, M., Calreticulin recognizes misfolded HLA-A2 heavy chains, Proc. Natl. Acad, Sci. USA, 99, 5931-5936 (2002).
Martal, J., L'embryon chez l'homme et l'animal Science update (Paris), editions Quae, 203-211, ISBN 2738009670, 9782738009678 (2002).
Matsuo, M, Asakawa, S, Shimizu, N, Kimura, H, Nonaka, M. Nucleotide sequence of the MHC class I genomic region of a teleost, the medaka (Oryzias latipes). Immunogenetics. 53, 930-940 (2002).
Michaëlsson ,J., Achour, A., Rolle, A., Karre, K., MHC class I recognition by NK receptors in the Ly49 family is strongly influenced by the beta 2-microglobulin subunit, J. Immunol., 166, 7327-7334 (2001).
Michalova, V., Murray, B.W., Sultmann, H., Klein, J., A contig map of the Mhc class I genomic region in the zebrafish reveals ancient synteny, J Immunol., 164, 5296-5305 (2000).
Miley, M.J., Truscott, S.M., Yu, Y.Y.L, Gilfillan, S., Fremont, D.H., Hansen, T.H, Lybarger, L., Biochemical Features of the MHC-Related Protein 1 Consistent with an Immunological Function, J. Immunol., 170, 6090-6098 (2003).
Mosyak, L., Zaller, D.M., Wiley, D.C., The structure of HLA-DM, the peptide exchange catalyst that loads antigen onto class II MHC molecules during antigen presentation, Immunity, 9, 377-383 (1998).
Neefjes, J.J., Hämmerling, G.J., Momburg, F., Folding and assembly of major histocompatibility complex class I heterodimers in the endoplasmic reticulum of intact cells precedes the binding of peptide, J. Exp. Med., 178, 1971-1980 (1993).
Ortmann, B., Androlewicz, M.J., Cresswell, P., MHC class I/beta2-microglobulin complexes associate with TAP transporters before peptide binding, Nature, 368, 864-867 (1994).
Phillips, RB., Zimmerman, A., Noakes, M.A., Palti, Y., Morasch, M.R., Eiben, L., Ristow, S.S., Thorgaard, G.H., Hansen, J.D., Physical and genetic mapping of the rainbow trout major histocompatibility regions: evidence for duplication of the class I region. Immunogenetics, 55, 561-569 (2003).
Rammensee, H. G., Chemistry of peptides associated with MHC class I and class II molecules, Curr Opin Immunol., 7(1), 85-96 (1995).
Rodrigues, P.N.S., Dixon, B., Roelofs ,J., Rombout, J.H.M.W., Egberts, E., Pohajdak, B., Stet, R.J.M., Expression and temperaturedependent regulation of the beta2-microglobulin (Cyca-B2m) gene in a cold-blooded vertebrate, the common carp (Cyprinus carpio L.), Dev Immunol., 5, 263-275 (1998).
Sadasivan, B., Lehner, P.J., Ortmann, B., Spies, T., Cresswell, P., Roles for calreticulin and a novel glycoprotein, tapasin, in the interaction of MHC class I molecules with TAP, Immunity, 5, 103-114 (1996).
Sambrook, J.G., Figueroa, F, Beck, S., A genome-wide survey of Major Histocompatibility Complex (MHC) genes and their paralogues in zebrafish, BMC Genomics, 6, 152-162 (2005).
Sánchez, L.M., Chirino, A.J., Bjorkman, P.J., Crystal Structure of Human ZAG, a Fat-Depleting Factor Related to MHC Molecules, Science, 283, 1914-1919 (1999).
Saper, M.A., Bjorkman, J.P., Wiley, D.C., Refined structure of the human histocompatibility antigen HLA-A2 at 2.6 A resolution, J. Mol. Bio., 219(2), 277-319 (1991).
Schleinitz, N., Dignat-George, F., Sampol, J., et al., Les cellules naturalkiller, Revue française des laboratoires, 341, 23-30 (2002).
Sege, K., Rask, L., Peterson, P.A., Role of b2-microglobulin in the intracellular processing of HLA antigens, Biochemistry, 20, 4523-4530 (1981).
Shiina, T, Inoko, H, Kulski, JK., An update of the HLA genomic region, locus information and disease associations, Tissue Antigens, 64, 631-649 (2004).
Shiina, T., Martinus, Dijkstra, J., Shimizu, S., Watanabe, A., Yanagiya, K., Kiryu, I., Fujiwara, A., Nishida-Umehara, C., Kaba, Y., Hirono, I., Yoshiura, Y., Aoki, T., Inoko, H., Kazimierz Kulski, J., Ototake, M., Interchromosomal duplication of major histocompatibility complex class I regions in rainbow trout (Oncorhynchus mykiss),a species with a presumably recent tetraploid ancestry, Immunogenetics, 56, 878-893 (2005).
Simmonds, R.E., Lane, D.A., Structural and functional implications of the intron/exon organization of the human endothelial cell protein C/activated protein C receptor (EPCR) gene: comparison with the structure of CD1/major histocompatibility complex alpha1 and alpha2 domains, Blood, 94(2), 632-41 (1999).
Smith, J.D., Solheim, J.C., Carreno, B.M., Hansen, T.H., Characterization of class I MHC folding intermediates and their disparate interactions with peptide and b2-microglobulin, Mol. Immunol., 32, 531-540 (1995).
Snyder, G.A., Brooks, A.G., Sun, P.D., Crystal structure of the HLA-Cw3 allotype-specific killer cell inhibitory receptor KIR2DL2, Proc. Natl. Acad. Sci. USA, 96, 3864-3869 (1999).
Stewart-Jones, G.B.E., McMichael, A.J., Bell, J.I., Stuart, D.I., Jones, E.Y., A structural basis for immunodominant human T cell receptor recognition, Nature Immunol., 4, 657-663 (2003).
Teng, M.K., Smolyar, A., Tse, A.G.D., Liu, J.H., Liu, J., Hussey, R.E., Nathenson, S.G., Chang, H.C., Reinherz, E.L., Wang, J.H., Identification of a common docking topology with substantial variation among different TCR-peptide-MHC complexes, Curr Biol, 8, 409-412 (1998).
Tormo, J., Natarajan, K., Margulies, D.H., Mariuzza, R.A., Crystal structure of a lectin-like natural killer cell receptor bound to its MHC class I ligand, Nature, 402, 623-631 (1999).
Townsend, A., Elliot, T., Cerundolo, V., Foster, L., Barber, B., Tse, A., Assembly of MHC class I molecules analyzed in vitro, Cell, 62, 285-295 (1990).
Trinchieri, G., Biology of natural killer cells, Adv. Immunol., 47, 187-376 (1989).
Tsukamoto, K., Hayashi, S., Matsuo, M., Nonaka, M., Kondo, M., Shima, M.I., Asakawa, S., Shimizu, N., Nonaka, M., Unprecedented intraspecific diversity of the MHC class I region of a teleost medaka, Oryzias latipes, Immunogenetics, 57, 420-431 (2005).
Uebel, S., Tampé, R., Specificity of the proteasome and the TAP transporter, Curr. Opin. Immunol., 11, 203-208 (1999).
Van Lith, M., Van Ham, M., Neefjes, J., Stable expression of MHC class I heavy chain/HLA-DO complexes at the plasma membrane, Eur. J. Immunol., 33, 1145-1151 (2003).
Vasmatzis, G., Cornette, J., Sezerman, U., DeLisi, C., TcR Recognition of the MHC-Peptide Dimer: Structural Properties of a Ternary Complex, Journal of Molecular Biology, 261, 72-89 (1996).
Villadangos, J.A., Ploegh, H.L., Proteolysis in MHC class II antigen presentation: who's in charge?, Immunity, 12, 233-239 (2000).
West, A.P., Bjorkman, P.J., Crystal structure and immunoglobulin G binding properties of the human major histocompatibility complex-related Fc receptor, Biochemistry, 39(32), 9698-708 (2000).
Willcox, B.E., Thomas, L.M., Bjorkman, P.J., Crystal structure of HLA-A2 bound to LIR-1, a host and viral major histocompatibility complex receptor, Nature Immunol., 4, 913-919, (2003).
Xiaohua Wei ; Orr H. T.; Differential expression of HLA-E, HLA-F, and HLA-G transcripts in human tissue 29(2), 131-142 (1990).
Yewdell, J.W., Reits, E., Neefjes, J., Making sense of mass destruction: quantitating MHC class I antigen presentation, Nature Rev. Immunol., (3), 952-961 (2003).
Yokoyama, W.M., HLA class I specificity for natural killer cell receptor CD94/NKG2A: two for one in more ways than one, Proc. Natl. Acad. Sci., 95, 4791-4794 (1998).
Zeng, Z.H., Castaño, A.R., Segelke, B.W., Stura, E.A., Peterson, P.A., Wilson, I.A., Crystal Structure of Mouse CD1: An MHC-Like Fold with a Large Hydrophobic Binding Groove, Science, 277(5324), 339-345 (1997).
Zhu, Y., Rudensky, A.Y., Corper, A.L., Teyton, L., Wilson, I.A., Crystal structure of MHC class II I-Ab in complex with a human CLIP peptide: prediction of an I-Ab peptide-binding motif, J. Mol. Biol., 326, 1157-1174 (2003).



